GUS się wychylił niespodziewanie z dużą paczką danych nt. zgonów. Dane są tygodniowe, w podziale na płcie, regiony w klasyfikacji NUTS oraz 5 letnie grupy wiekowe.
Dane są udostępnione w formacie XSLX w dość niepraktycznej z punktu widzenia przetwarzania strukturze (kolumny to tygodnie/wiersze to różne kategorie agregacji: płeć, wiek, region), który zamieniłem na CSV o następującej prostej 7 kolumnowej strukturze:
year;sex;week;date;age;geo;value
W miarę oczywiste jest, że year to rok, sex to płeć, week to numer tygodnia, date to data pierwszego dnia tygodnia (poniedziałek), geo to identyfikator obszaru a value liczba zgonów odpowiadająca kolumn 1--6. Ten plik jest podzielony na lata bo w całości zajmuje circa 200Mb. Umieściłem go tutaj.
Skrypt też w R wymodziłem co wizualizuje zgony wg grup wieku oraz województw. Ponieważ kombinacji płeć/wiek/region są setki, moje wykresy dotyczą zgonów ogółem/kobiet/mężczyzn w podziale na grupy wiekowe oraz ogółem w podziale na województwa. Każdy wykres zawiera dwa szeregi: liczbę zgonów w 2020 roku oraz średnią liczbę zgonów z lat 2015--2019. Ponadto jest wykres z jedną krzywą: procent liczony dla stosownych tygodni jako liczba zgonów w 2020 przez średnią 5 letnią z lat 2015--2019. Ten wykres występuje też w wariancie skróconym: tylko 6 ostatnich tygodni, co pozwala dodać do punktów wartości liczbowe (które nie zachodzą na siebie).
library("ggplot2")
library("dplyr")
library("scales")
library("ggthemes")
library("ggpubr")
library("tidyr")
picWd <- 12
spanV <- 0.5
GUS.url <- "https://stat.gov.pl/obszary-tematyczne/ludnosc/ludnosc/zgony-wedlug-tygodni,39,2.html"
NIKW.url <- "(c) NI-KW @ github.com/knsm-psw/GUS_mortality"
NIKW <- sprintf ("%s | %s", GUS, NIKW.url)
z <- read.csv("PL-mortality-2015.csv", sep = ';', header=T, na.string="NA" )
lastO <- last(z$date)
lastT <- last(z$week)
nuts <- c('PL21', 'PL22', 'PL41', 'PL42', 'PL43', 'PL51', 'PL52', 'PL61', 'PL62',
'PL63', 'PL71', 'PL72', 'PL81', 'PL82', 'PL84', 'PL91', 'PL92')
### Ogółem
z00 <- z %>% filter ( sex == 'O' & geo == 'PL' ) %>% as.data.frame
z0 <- z00 %>% filter ( year >= 2015 & year < 2020 ) %>% as.data.frame
z1 <- z00 %>% filter ( year == 2020 ) %>% as.data.frame
## średnie w okresie 1 -- (n-1)
zz0 <- z0 %>% group_by(age,week) %>% summarise( year = 't19',
vv = mean(value, na.rm=TRUE)) %>% as.data.frame
zz1 <- z1 %>% group_by(age,week) %>% summarise( year = 't20',
vv = mean(value, na.rm=TRUE)) %>% as.data.frame
### Połącz
zz1 <- bind_rows(zz0, zz1)
farbe19 <- '#F8766D'
farbe20 <- '#00BFC4'
p1 <- ggplot(zz1, aes(x=week, y=vv, color=year)) +
geom_smooth(method="loess", se=F, span=spanV, size=.4) +
geom_point(size=.4, alpha=.5) +
facet_wrap( ~age, scales = "free_y") +
xlab(label="") +
ylab(label="") +
##theme_nikw()+
theme(plot.subtitle=element_text(size=9), legend.position="top")+
scale_color_manual(name="Rok: ", labels = c("średnia 2015--2019", "2020"), values = c("t19"=farbe19, "t20"=farbe20 )) +
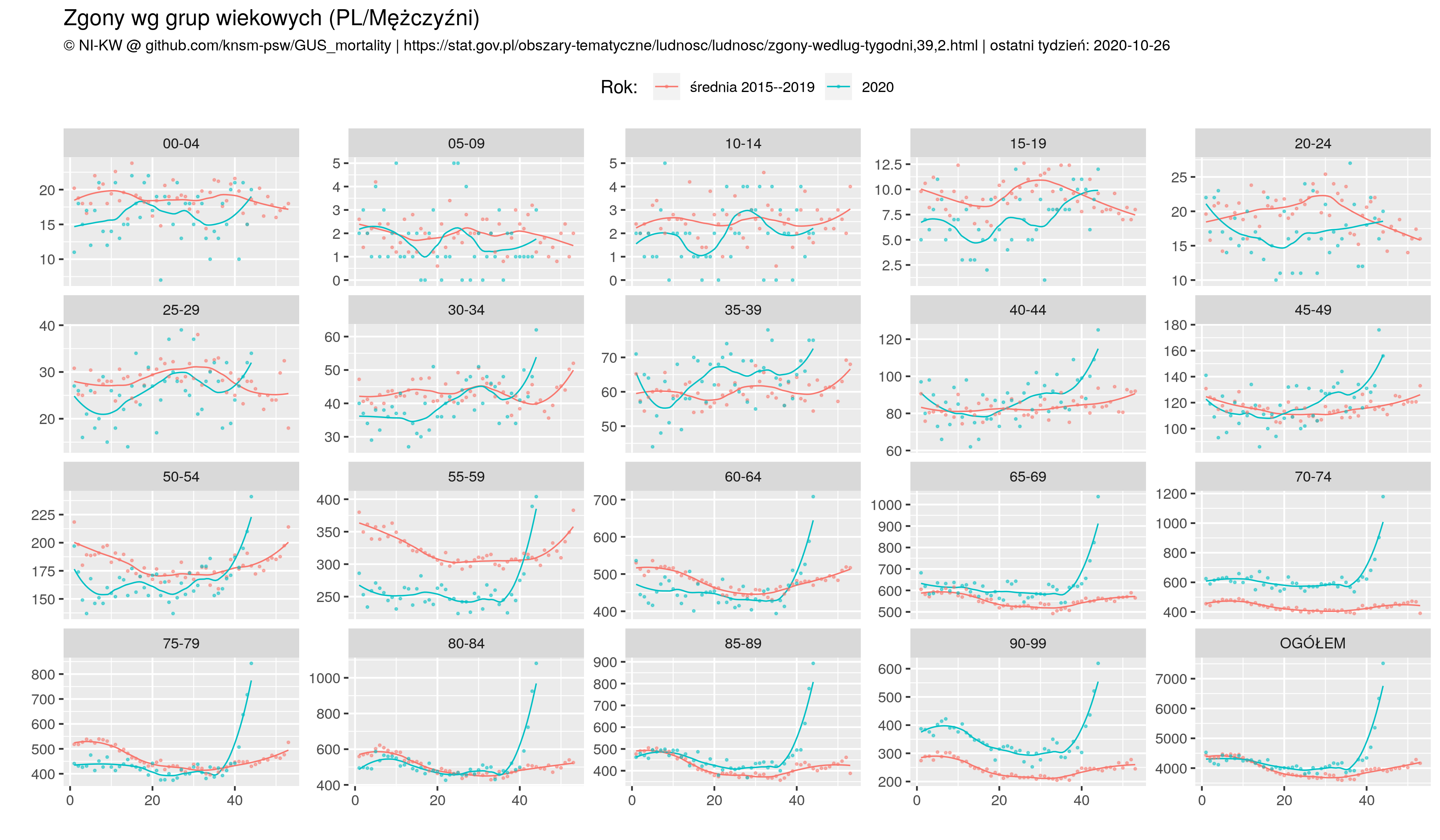
ggtitle("Zgony wg grup wiekowych (PL/Ogółem)", subtitle=sprintf("%s | ostatni tydzień: %s", NIKW, lastO) )
ggsave(plot=p1, "zgony_PL_by_age_O.png", width=picWd)
### M ###
z00 <- z %>% filter ( sex == 'M' & geo == 'PL' ) %>% as.data.frame
z0 <- z00 %>% filter ( year >= 2015 & year < 2020 ) %>% as.data.frame
z1 <- z00 %>% filter ( year == 2020 ) %>% as.data.frame
## średnie w okresie 1 -- (n-1)
zz0 <- z0 %>% group_by(age,week) %>% summarise( year = 't19',
vv = mean(value, na.rm=TRUE)) %>% as.data.frame
zz1 <- z1 %>% group_by(age,week) %>% summarise( year = 't20',
vv = mean(value, na.rm=TRUE)) %>% as.data.frame
### Połącz
zz1 <- bind_rows(zz0, zz1)
p2 <- ggplot(zz1, aes(x=week, y=vv, group=year, color=year)) +
geom_smooth(method="loess", se=F, span=spanV, size=.4) +
geom_point(size=.4, alpha=.5) +
facet_wrap( ~age, scales = "free_y") +
xlab(label="") +
ylab(label="") +
##theme_nikw()+
##labs(caption=source) +
theme(plot.subtitle=element_text(size=9), legend.position="top")+
scale_color_manual(name="Rok: ", labels = c("średnia 2015--2019", "2020"),
values = c("t19"=farbe19, "t20"=farbe20 )) +
ggtitle("Zgony wg grup wiekowych (PL/Mężczyźni)", subtitle=sprintf("%s | ostatni tydzień: %s", NIKW, lastO) )
ggsave(plot=p2, "zgony_PL_by_age_M.png", width=picWd)
### K #########################################
z00 <- z %>% filter ( sex == 'K' & geo == 'PL' ) %>% as.data.frame
z0 <- z00 %>% filter ( year >= 2015 & year < 2020 ) %>% as.data.frame
z1 <- z00 %>% filter ( year == 2020 ) %>% as.data.frame
## średnie w okresie 1 -- (n-1)
zz0 <- z0 %>% group_by(age,week) %>% summarise( year = 't19',
vv = mean(value, na.rm=TRUE)) %>% as.data.frame
zz1 <- z1 %>% group_by(age,week) %>% summarise( year = 't20',
vv = mean(value, na.rm=TRUE)) %>% as.data.frame
### Połącz
zz1 <- bind_rows(zz0, zz1)
p3 <- ggplot(zz1, aes(x=week, y=vv, group=year, color=year)) +
geom_smooth(method="loess", se=F, span=spanV, size=.4) +
geom_point(size=.4, alpha=.5) +
facet_wrap( ~age, scales = "free_y") +
xlab(label="") +
ylab(label="") +
##theme_nikw()+
##labs(caption=source) +
theme(plot.subtitle=element_text(size=9), legend.position="top")+
scale_color_manual(name="Rok: ", labels = c("średnia 2015--2019", "2020"),
values = c("t19"=farbe19, "t20"=farbe20 )) +
ggtitle("Zgony wg grup wiekowych (PL/Kobiety)", subtitle=sprintf("%s | ostatni tydzień: %s", NIKW, lastO) )
ggsave(plot=p3, "zgony_PL_by_age_K.png", width= picWd)
### ogółem wg województw #####################################
n <- read.csv("nuts.csv", sep = ';', header=T, na.string="NA" )
## dodaj nazwy
z <- left_join(z, n, by='geo')
## wiek razem
z00 <- z %>% filter ( sex == 'O' & geo %in% nuts & age == 'OGÓŁEM') %>% as.data.frame
z0 <- z00 %>% filter ( year >= 2015 & year < 2020 ) %>% as.data.frame
z1 <- z00 %>% filter ( year == 2020 ) %>% as.data.frame
## średnie w okresie 1 -- (n-1)
zz0 <- z0 %>% group_by(name,week) %>%
summarise( year = 't19', vv = mean(value, na.rm=TRUE)) %>% as.data.frame
zz1 <- z1 %>% group_by(name,week) %>%
summarise( year = 't20', vv = mean(value, na.rm=TRUE)) %>% as.data.frame
### Połącz
zz1 <- bind_rows(zz0, zz1)
lastWeek <- last(zz1$week)
firstWeek <- lastWeek - 6
zz1 <- zz1 %>% filter ( week >= firstWeek ) %>% as.data.frame
print(zz1)
p4 <- ggplot(zz1, aes(x=week, y=vv, group=year, color=year)) +
geom_smooth(method="loess", se=F, span=spanV, size=.4) +
geom_point(size=.4, alpha=.5) +
facet_wrap( ~name, scales = "free_y") +
xlab(label="") +
ylab(label="") +
##theme_nikw()+
##labs(caption=source) +
theme(plot.subtitle=element_text(size=9), legend.position="top")+
scale_color_manual(name="Rok: ", labels = c("średnia 2015--2019", "2020"),
values = c("t19"=farbe19, "t20"=farbe20 )) +
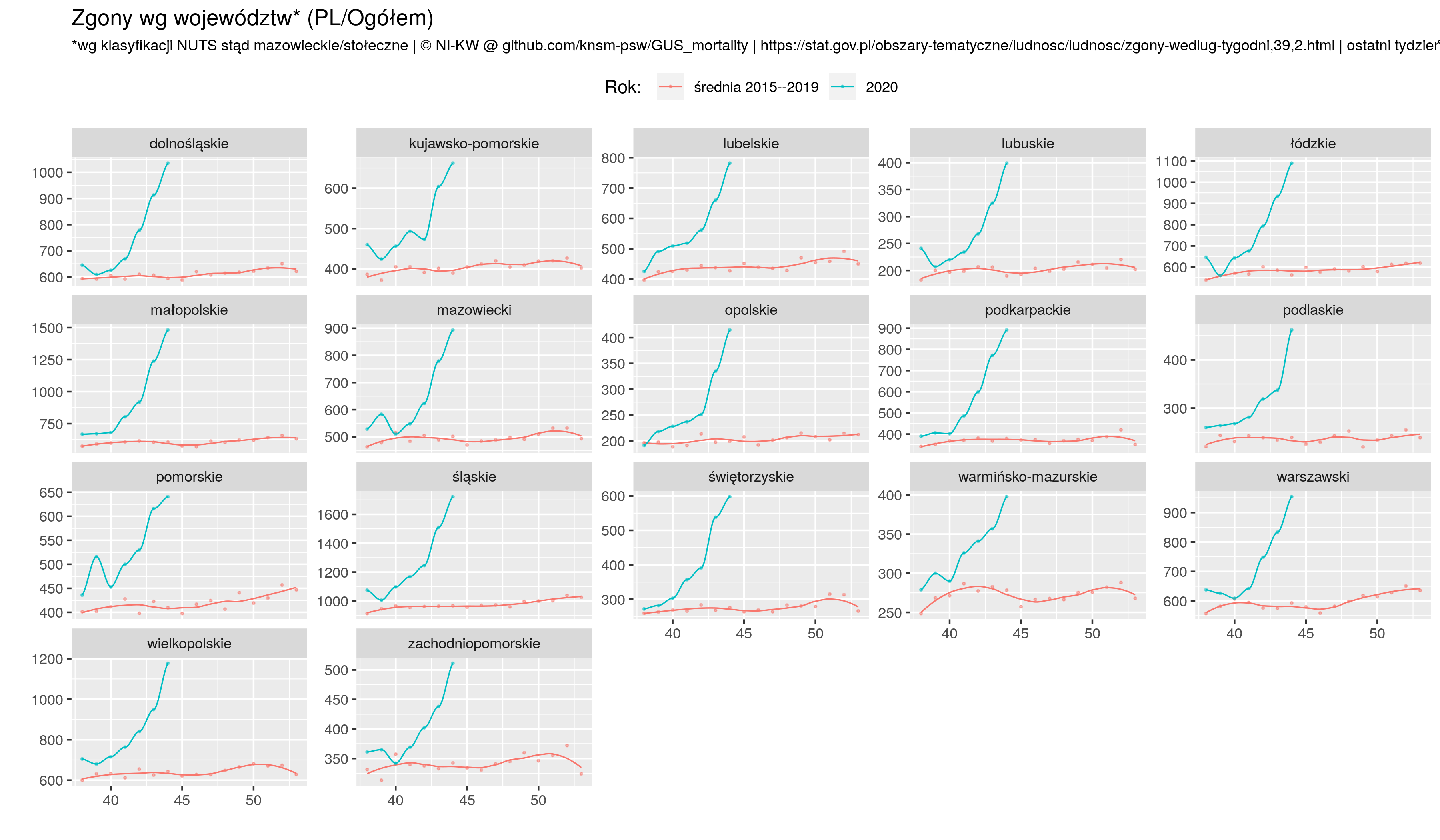
ggtitle("Zgony wg województw* (PL/Ogółem)",
subtitle=sprintf("*wg klasyfikacji NUTS stąd mazowieckie/stołeczne | %s | ostatni tydzień: %s", NIKW, lastO))
ggsave(plot=p4, "zgony_PL_by_woj_O.png", width=picWd)
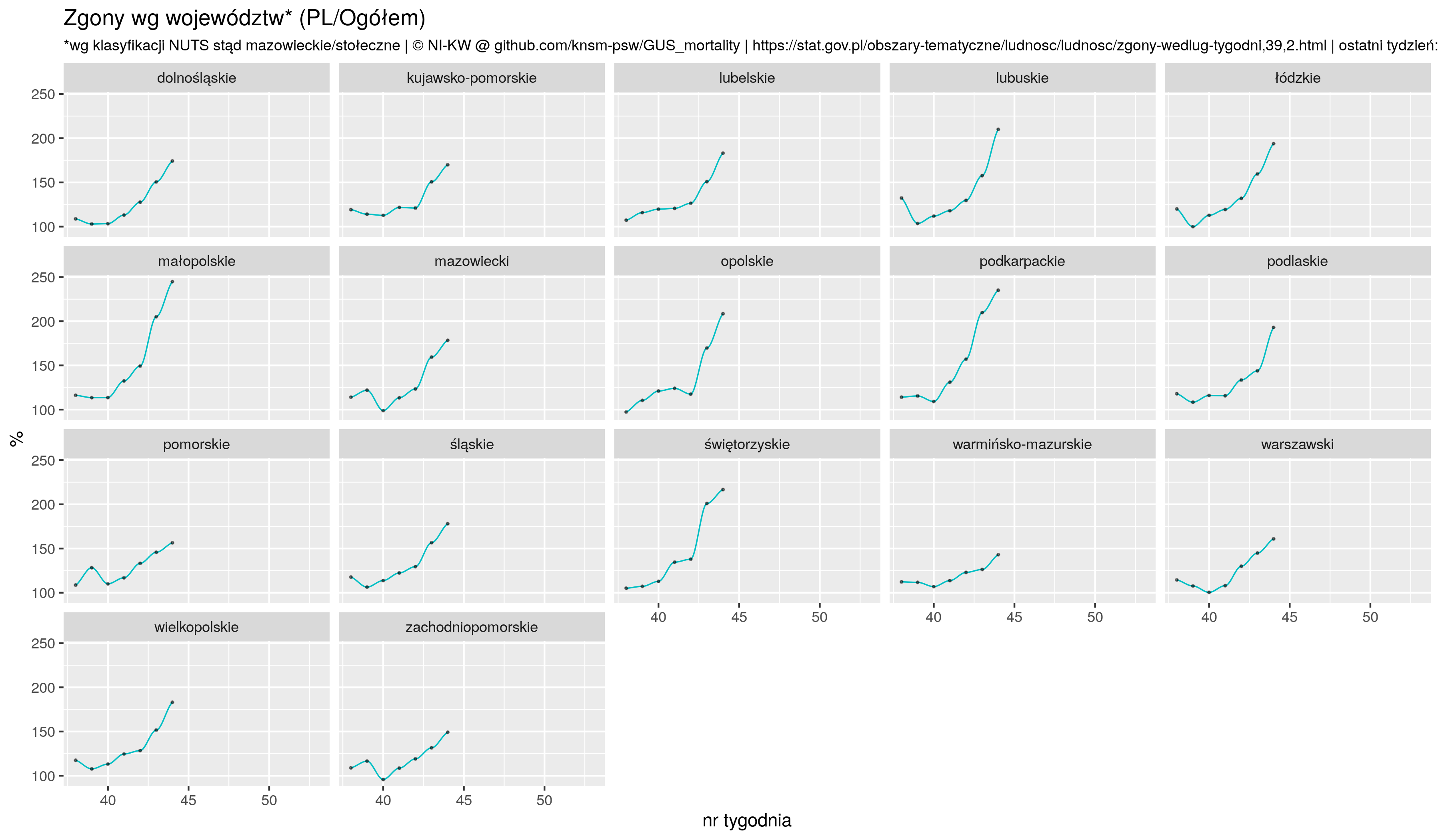
## jako %% w średniej w poprzednich 5 lat
zz1 <- zz1 %>% spread(year, vv)
zz1$yy <- zz1$t20 / zz1$t19 * 100
p5 <- ggplot(zz1, aes(x=week, y=yy), color=farbe20) +
geom_smooth(method="loess", se=F, span=spanV, size=.4, color=farbe20) +
geom_point(size=.4, alpha=.5) +
facet_wrap( ~name, scales = "fixed") +
xlab(label="nr tygodnia") +
ylab(label="%") +
theme(plot.subtitle=element_text(size=9), legend.position="top")+
scale_color_manual(name="Rok 2020: ", labels = c("% 2020/(średnia 2015--2015)"),
values = c("yy"=farbe20 ) ) +
ggtitle("Zgony wg województw* (PL/Ogółem)",
subtitle=sprintf("*wg klasyfikacji NUTS stąd mazowieckie/stołeczne | %s | ostatni tydzień: %s", NIKW, lastO))
ggsave(plot=p5, "zgony_PL_by_woj_P.png", width=picWd)
zz1 <- zz1 %>% filter ( week >= firstWeek ) %>% as.data.frame
p6 <- ggplot(zz1, aes(x=week, y=yy), color=farbe20) +
geom_smooth(method="loess", se=F, span=spanV, size=.4, color=farbe20) +
geom_point(size=.4, alpha=.5) +
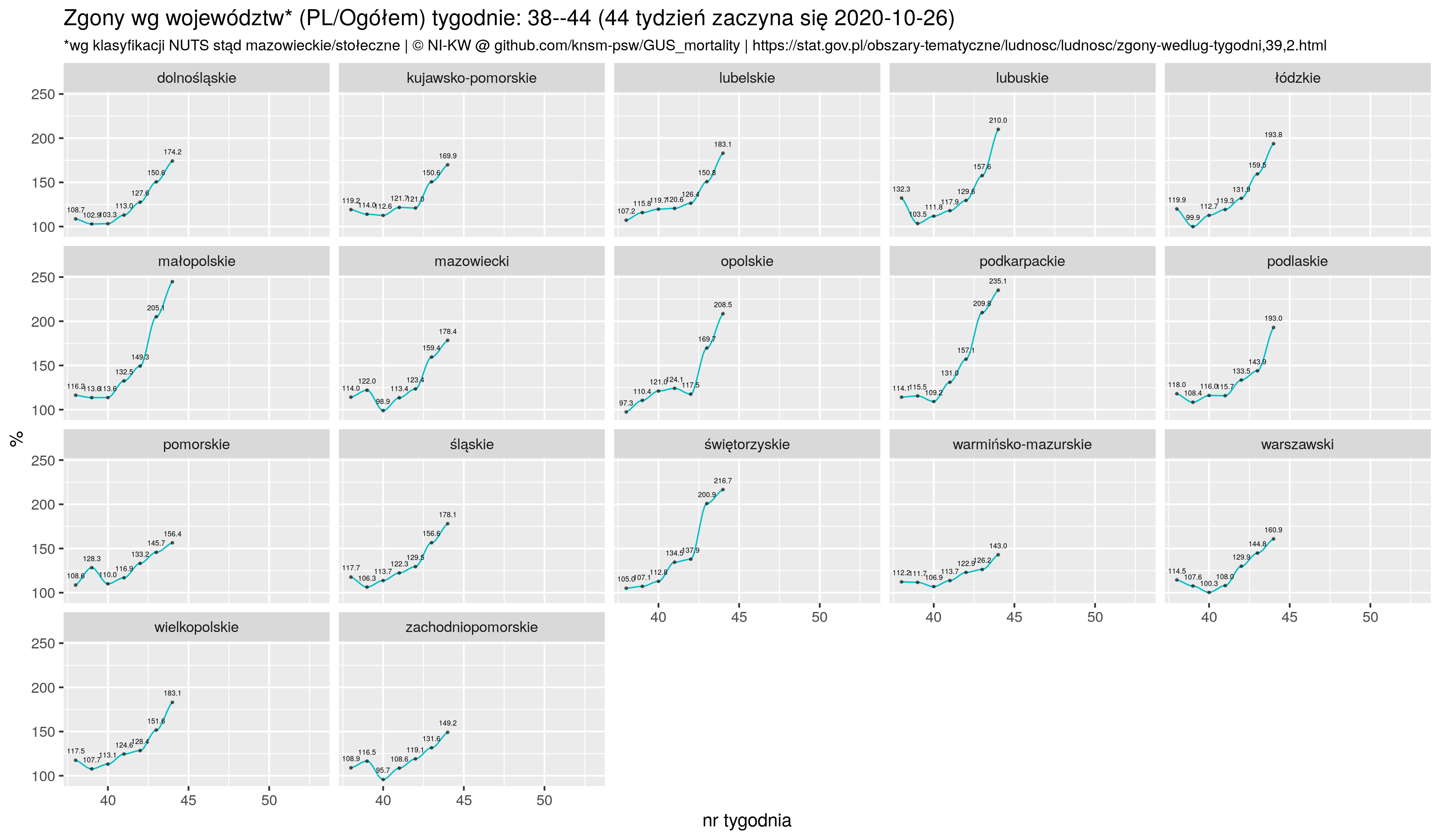
geom_text(aes(label=sprintf("%.1f", yy)), vjust=-1.25, size=1.5) +
facet_wrap( ~name, scales = "fixed") +
xlab(label="nr tygodnia") +
ylab(label="%") +
theme(plot.subtitle=element_text(size=9), legend.position="top")+
scale_color_manual(name="Rok 2020: ", labels = c("% 2020/(średnia 2015--2015)"),
values = c("yy"=farbe20 ) ) +
ggtitle(sprintf("Zgony wg województw* (PL/Ogółem) tygodnie: %i--%i (%i tydzień zaczyna się %s)",
firstWeek, lastWeek, lastWeek, lastO),
subtitle=sprintf("*wg klasyfikacji NUTS stąd mazowieckie/stołeczne | %s", NIKW))
ggsave(plot=p6, "zgony_PL_by_woj_P6.png", width=picWd)


Fascynujący wpis. Podoba mi się.
OdpowiedzUsuń