Wymyśliłem sobie wykreślić wykresy pokazujące zależność pomiędzy liczbą zarażonych/zmarłych, a wybranymi wskaźnikami: GDP (zamożność), oczekiwana długość życia (poziom służby zdrowia) oraz śmiertelność dzieci (zamożność/poziom rozwoju). Większość danych pobrałem z portalu OWiD:
- GDP (https://ourworldindata.org/economic-growth maddison-data-gdp-per-capita-in-2011us.csv)
- LE (https://ourworldindata.org/life-expectancy life-expectancy.csv)
- CM (https://ourworldindata.org/child-mortality child-mortality-igme.csv).
Dane dotyczące liczby ludności są z bazy Banku Światowego (https://data.worldbank.org/indicator/sp.pop.totl.) Dane dotyczące zarażonych/zmarłych ze strony ECDC (www.ecdc.europa.eu/en/covid-19/data-collection) Scaliłem wszystko do kupy skryptem Perlowym. NB pojawił się tzw. small problem, bo bazy OWiD oraz WorldBank używają ISO-kodów 3-literowych krajów, a ECDC dwuliterowych (PL vs POL). Trzeba było znaleźć wspólny mianownik. Szczęśliwie Perl ma gotowy moduł. Oprócz tego ECDC stosuje EU-standard w przypadku Grecji (EL zamiast GR) oraz Wielkiej Brytanii (UK/GB).
#!/usr/bin/perl
use Locale::Codes::Country;
...
while (<COVID>) { chomp();
($date, $iso2, $country, $newc, $newd, $totalc, $totald) = split /;/, $_;
$iso3 = uc ( country_code2code($iso2, 'alpha-2', 'alpha-3'));
}
Rezultat jest zapisywany do pliku o następującej zawartości:
iso3;country;lex2019;gdp2016;cm2017;pop2018;cases;deaths ABW;Aruba;76.29;NA;NA;105845;77;0 AFG;Afghanistan;64.83;1929;6.79;37172386;423;14 ...
Wierszy jest 204, przy czym niektórym krajom brakuje wartości. Ponieważ dotyczy to krajów egzotycznych, zwykle małych, to pominę je (nie teraz później, na etapie przetwarzania R-em). Takich wybrakowanych krajów jest 49 (można grep-em sprawdzić). Jedynym większym w tej grupie jest Syria, ale ona odpada z innych powodów.
Do wizualizacji wykorzystam wykres punktowy (dot-plot) oraz wykresy rozrzutu (dot-plot).
library("dplyr")
library("ggplot2")
library("ggpubr")
##
options(scipen=1000000)
## https://www.r-bloggers.com/the-notin-operator/
`%notin%` <- Negate(`%in%`)
##
today <- Sys.Date()
tt<- format(today, "%d/%m/%Y")
million <- 1000000
## Lista krajów Europejskich + Izrael
## (pomijamy kraje-liliputy)
ee <- c(
'BEL', 'GRC', 'LTU', 'PRT', 'BGR', 'ESP', 'LUX', 'ROU', 'CZE', 'FRA', 'HUN',
'SVN', 'DNK', 'HRV', 'MLT', 'SVK', 'DEU', 'ITA', 'NLD', 'FIN', 'EST', 'CYP',
'AUT', 'SWE', 'IRL', 'LVA', 'POL', 'ISL', 'NOR', 'LIE', 'CHE', 'MNE', 'MKD',
'ALB', 'SRB', 'TUR', 'BIH', 'BLR', 'MDA', 'UKR', 'ISR', 'RUS', 'GBR' );
ee.ee <- c('POL')
d <- read.csv("indcs.csv", sep = ';', header=T, na.string="NA");
## Liczba krajów
N1 <- nrow(d)
## liczba ludności w milionach
d$popm <- d$pop / million
## Oblicz współczynniki na 1mln
d$casesr <- d$cases/d$popm
d$deathsr <- d$deaths/d$popm
## Tylko kraje wykazujące zmarłych
d <- d %>% filter(deaths > 0) %>% as.data.frame
## Liczba krajów wykazujących zmarłych
N1d <- nrow(d)
## Tylko kraje z kompletem wskaźników (pomijamy te z brakami)
d <- d[complete.cases(d), ]
nrow(d)
## UWAGA: pomijamy kraje o wsp. <= 2
## droplevels() usuwa `nieużywane' czynniki
## mutate zmienia kolejność na kolejność wg dearhsr:
d9 <- d %>% filter(deathsr > 2 ) %>% droplevels() %>%
mutate (iso3 = reorder(iso3, deathsr)) %>% as.data.frame
N1d2 <- nrow(d9)
M1d2 <- median(d9$deathsr, na.rm=T)
## https://stackoverflow.com/questions/11093248/geom-vline-with-character-xintercept
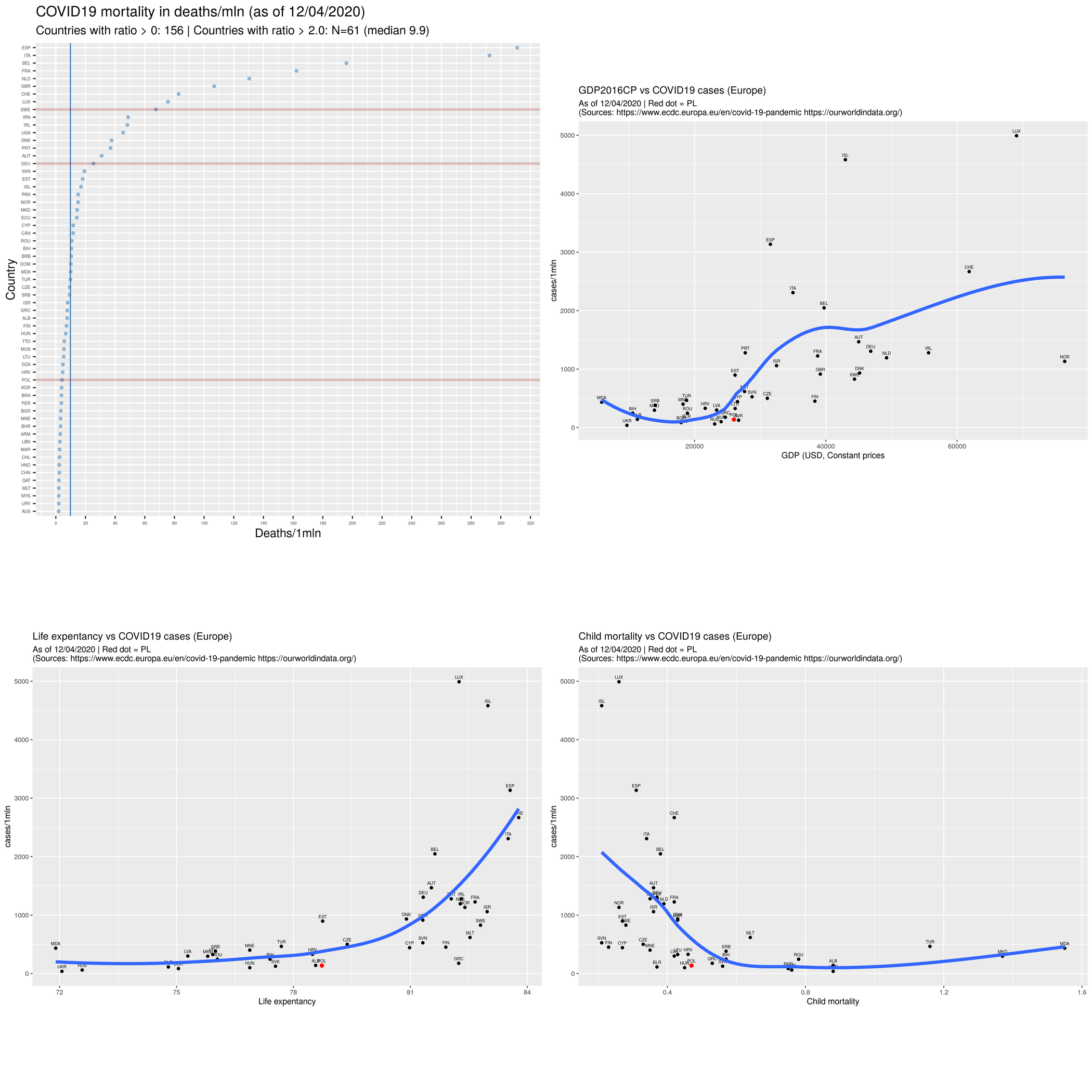
## Wykres punktowy
rys99 <- ggplot(d9, aes(x =iso3, y = deathsr )) +
geom_point(size=1, colour = 'steelblue', alpha=.5) +
xlab(label="Country") +
ylab(label="Deaths/1mln") +
ggtitle(sprintf("COVID19 mortality in deaths/mln (as of %s)", tt),
subtitle=sprintf("Countries with ratio > 0: %i | Countries with ratio > 2.0: N=%i (median %.1f)",
N1d, N1d2, M1d2)) +
theme(axis.text = element_text(size = 4)) +
##theme(plot.title = element_text(hjust = 0.5)) +
scale_y_continuous(breaks=c(0,20,40,60,80,100,120,140,160,180,200,220,240,260,280,300,320,340,360)) +
geom_hline(yintercept=M1d2, linetype="solid", color = "steelblue") +
geom_vline(aes(xintercept = which(levels(iso3) == 'POL')), size=1, color="#8A0303", alpha=.25) +
geom_vline(aes(xintercept = which(levels(iso3) == 'DEU')), size=1, color="#8A0303", alpha=.25) +
geom_vline(aes(xintercept = which(levels(iso3) == 'SWE')), size=1, color="#8A0303", alpha=.25) +
coord_flip()
Uwaga: oś OX to skala porządkowa. Czynniki powinny być uporządkowane wg. wartości zmiennej z osi OY, czyli według deathsr. Do tego służy funkcja mutate (iso3 = reorder(iso3, deathsr). Można też uporządkować je ,,w locie'' aes(x =iso3, y = deathsr ), ale wtedy niepoprawnie będą kreślone (za pomocą geom_vline) linie pionowe. Linie pionowe służą do wyróżnienia pewnych krajów. Linia pozioma to linia mediany.
sources <- sprintf ("As of %s\n(Sources: %s %s)", tt,
"https://www.ecdc.europa.eu/en/covid-19-pandemic",
"https://ourworldindata.org/")
## Żeby etykiety nie zachodziły na siebie tylko dla wybranych krajów
## Add empty factor level! Istotne inaczej będzie błąd
# https://rpubs.com/Mentors_Ubiqum/Add_Levels
d$iso3 <- factor(d$iso3, levels = c(levels(d$iso3), ""))
d$iso3xgdp <- d$iso3
d$iso3xlex <- d$iso3
d$iso3xcm <- d$iso3
## Kraje o niskich wartościach bez etykiet
## Bez etykiet jeżeli GDP<=45 tys oraz wskaźnik < 50:
d$iso3xgdp[ (d$gdp2016 < 45000) & (d$deathsr < 50 ) ] <- ""
## Inne podobnie
d$iso3xlex[ ( (d$lex2019 < 80) | (d$deathsr < 50 ) ) ] <- ""
d$iso3xcm[ ((d$cm2017 > 1.2) | (d$deathsr < 50 ) ) ] <- ""
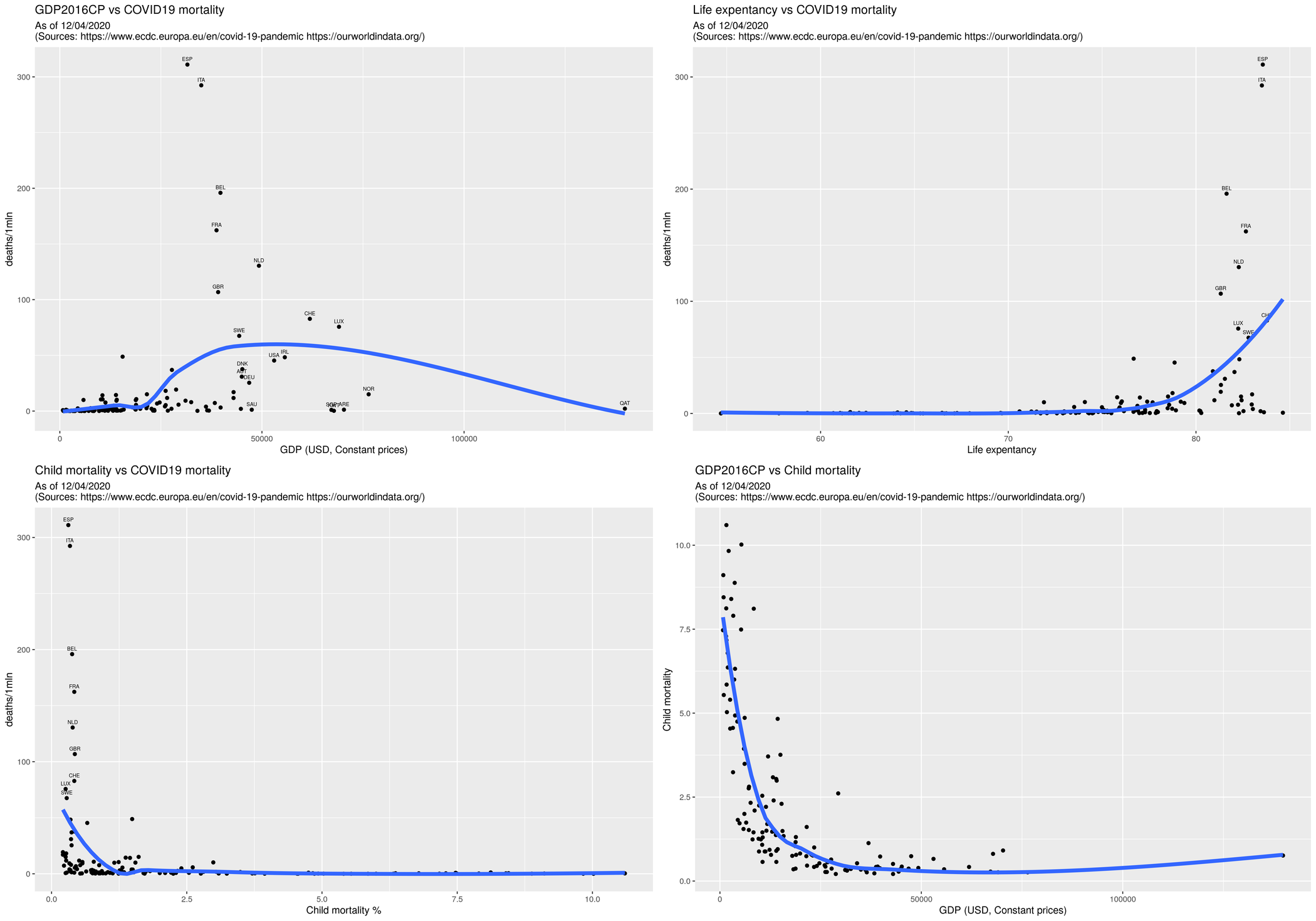
## GDP vs współczynnik zgonów/1mln
rys1 <- ggplot(d, aes(x=gdp2016, y=deathsr)) +
geom_point() +
geom_text(data=d, aes(label=sprintf("%s", iso3xgdp), x=gdp2016, y= deathsr), vjust=-0.9, size=2 ) +
xlab("GDP (USD, Constant prices)") +
ylab("deaths/1mln") +
geom_smooth(method="loess", se=F, size=2) +
ggtitle("GDP2016CP vs COVID19 mortality", subtitle=sources)
## Life ex vs współczynnik zgonów/1mln
rys2 <- ggplot(d, aes(x=lex2019, y=deathsr)) +
geom_point() +
geom_text(data=d, aes(label=sprintf("%s", iso3xlex), x=lex2019, y= deathsr), vjust=-0.9, size=2 ) +
xlab("Life expentancy") +
ylab("deaths/1mln") +
geom_smooth(method="loess", se=F, size=2) +
ggtitle("Life expentancy vs COVID19 mortality", subtitle=sources)
## Child mortality vs współczynnik zgonów/1mln
rys3 <- ggplot(d, aes(x=cm2017, y=deathsr)) +
geom_point() +
geom_text(data=d, aes(label=sprintf("%s", iso3xcm), x=cm2017, y= deathsr), vjust=-0.9, size=2 ) +
xlab("Child mortality %") +
ylab("deaths/1mln") +
geom_smooth(method="loess", se=F, size=2) +
ggtitle("Child mortality vs COVID19 mortality", subtitle=sources)
## GDP vs Child mortality
rys0 <- ggplot(d, aes(x=gdp2016, y=cm2017)) +
geom_point() +
xlab("GDP (USD, Constant prices)") +
ylab("Child mortality") +
geom_smooth(method="loess", se=F, size=2) +
ggtitle("GDP2016CP vs Child mortality", subtitle=sources)
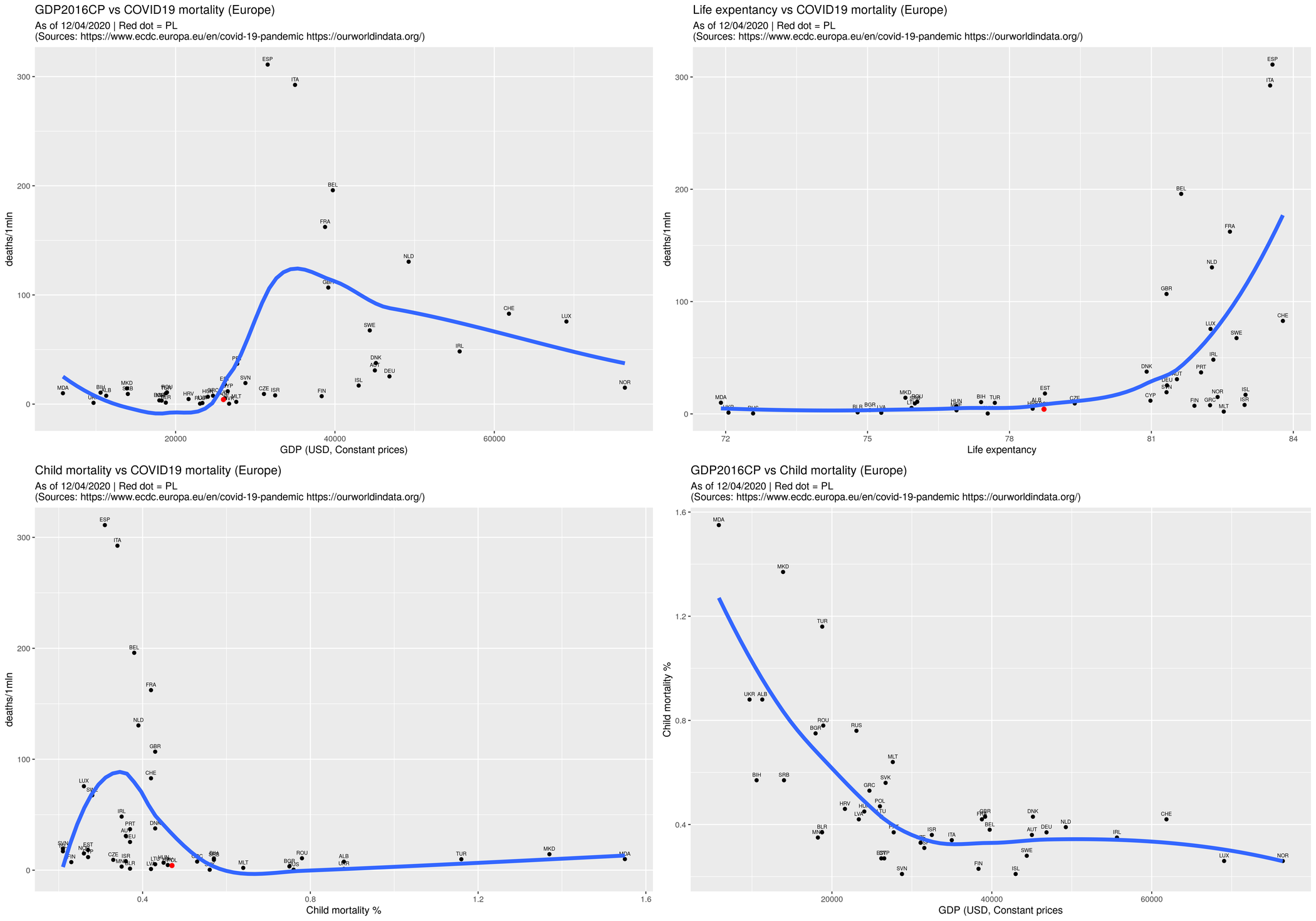
Jeszcze raz dla krajów Europejskich:
## Tylko kraje Europejskie:
d <- d %>% filter (iso3 %in% ee) %>% as.data.frame
d$iso3xgdp <- d$gdp2016
d$iso3xlex <- d$lex2019
d$iso3xcm <- d$cm2017
d$iso3xgdp[ d$iso3 %notin% ee.ee ] <- NA
d$iso3xlex[ d$iso3 %notin% ee.ee ] <- NA
d$iso3xcm[ d$iso3 %notin% ee.ee ] <- NA
rys1ec <- ggplot(d, aes(x=gdp2016, y=casesr)) +
geom_point() +
geom_text(data=d, aes(label=sprintf("%s", iso3), x=gdp2016, y= casesr), vjust=-0.9, size=2 ) +
geom_point(data=d, aes(x=iso3xgdp, y= casesr), size=2, color="red" ) +
xlab("GDP (USD, Constant prices") +
ylab("cases/1mln") +
geom_smooth(method="loess", se=F, size=2) +
ggtitle("GDP2016CP vs COVID19 cases (Europe)", subtitle=sources)
... itd...
Wynik w postaci rysunków:
Powyższe jest dostępne tutaj
Brak komentarzy:
Prześlij komentarz