Jeszcze jeden skrypt do rysowania danych nt COVID19:
#!/usr/bin/env Rscript
# Przekazywanie argumentów z wiersza poleceń
# np.: Rscript --vanilla c19.R --iso PL -clean
library("optparse")
#
library("ggplot2")
library("dplyr")
library("scales")
library("ggpubr")
#
# parametr wygładzania (loess)
spanV <- 0.25
# UWAGA: przed/po \n musi być odstęp inaczej nie działa
surl <- "https://www.ecdc.europa.eu/en/publications-data/ \n download-todays-data-geographic-distribution-covid-19-cases-worldwide"
c0 <- 'PL'
option_list <- list(
make_option(c("-i", "--iso"), action="store", type="character", default=c0, help="country ISO2 code"),
make_option(c("-c", "--clean"), action="store_true", default=T, help="extra clean data?")
);
opt_parser <- OptionParser(option_list=option_list);
opt <- parse_args(opt_parser);
c0 <- opt$iso
dataClean <- opt$clean
# wczytanie danych
# date;id;country;newc;newd;totalc;totald
d <- read.csv("covid19_C.csv", sep = ';', header=T, na.string="NA",
colClasses = c('factor', 'factor', 'factor', 'character', 'character', 'numeric', 'numeric'));
str(d)
d$newc <- as.numeric(d$newc)
d$newd <- as.numeric(d$newd)
## Dane zawierają wartości ujemne co jest bez sensu
## z opcją --clean te ujemne wartości są zamieniane na NA
if ( dataClean ) {
cat ("### Cleaning newc/newd: assign NA to negatives...\n")
d$newc[ (d$newc < 0) ] <- NA
d$newd[ (d$newd < 0) ] <- NA
}
## Współczynniki zmarli/zakażeni
d$newr <- d$newd/d$newc * 100
d$totr <- d$totald/d$totalc * 100
## Wartości > 50% są zamieniane na NA (zwykle >50% wynika z błędnych danych)
if ( dataClean ) {
cat ("### Cleaning newc/newd: assign NA to newr/totr higher than 50...\n")
d$newr[ (d$newr > 50) ] <- NA
d$totr[ (d$totr > 50) ] <- NA
}
## Pomiń obserwacje wcześniejsze niż 15/02
d <- d %>% filter(as.Date(date, format="%Y-%m-%d") > "2020-02-15") %>% as.data.frame
d0 <- d %>% filter (id == c0) %>% as.data.frame
t0 <- d0 %>% group_by(id) %>% summarise(cc = sum(newc, na.rm=T), dd=sum(newd, na.rm=T))
lab0c <- toString(paste (sep=" = ", t0$id, t0$cc))
lab0d <- toString(paste (sep=" = ", t0$id, t0$dd))
## koniecznie dodać na.rm=T bo inaczej zwraca NA (jeżeli znajdzie NA)
maxCC <- max (d0$newc, na.rm=T)
maxDD <- max (d0$newd, na.rm=T)
maxRR <- max (d0$totr, na.rm=T)
last.obs <- last(d0$date)
first.date <- first(d0$date)
fstDay <- as.Date(first.date)
last.totr <- last(d0$totr)
max.newr <- max(d0$newr, na.rm=T)
## Przykład dodania 14 dni do daty
## srcDay <- as.Date(first.date) +14
## https://stackoverflow.com/questions/10322035/r-adding-days-to-a-date
srcDay <- as.Date(last.obs)
## Nazwa pliku wynikowego
## c19_ISO2_DATA.png, gdzie DATA jest datą ostatniej obserwacji
## np.: c19_SE_2020-09-16.png
c0o <- sprintf ("c19_%s_%s.png", c0, last.obs)
## Rysunek1: nowe przypadki
pc0 <- ggplot(d0, aes(x= as.Date(date, format="%Y-%m-%d"), y=newc)) +
geom_point(aes(group = id, color = id), size=.8) +
geom_smooth(method="loess", se=F, span=spanV) +
theme(plot.subtitle=element_text(size=8, hjust=0, color="black")) +
scale_x_date( labels = date_format("%m/%d"), breaks = "2 weeks") +
annotate("text", x = fstDay, y = 0.95 * maxCC,
label = sprintf("Total: %i cases", t0$cc), hjust = 0, vjust=0,
alpha=0.3, color='steelblue', size=6) +
xlab(label="") +
## Nie drukuj legendy
theme(legend.position="none") +
ggtitle(sprintf("%s: new confirmed cases (%s)", t0$id, last.obs))
## Rysunek2: nowe zgony
pd0 <- ggplot(d0, aes(x= as.Date(date, format="%Y-%m-%d"), y=newd)) +
geom_point(aes(group = id, color = id), size=.8) +
geom_smooth(method="loess", se=F, span=spanV) +
theme(plot.subtitle=element_text(size=8, hjust=0, color="black")) +
annotate("text", x = fstDay, y = 0.95 * maxDD, label = sprintf("Total: %i deaths", t0$dd), hjust = 0, vjust=0,
alpha=0.3, color='steelblue', size=6) +
scale_x_date( labels = date_format("%m/%d"), breaks = "2 weeks") +
xlab(label="") +
theme(legend.position="none") +
ggtitle(sprintf ("%s: deaths (%s)", t0$id, last.obs))
## Rysunek3: nowe zgony/przypadki *100%
pr0 <- ggplot(d0, aes(x= as.Date(date, format="%Y-%m-%d"), y=newr)) +
geom_point(aes(group = id, color = id), size=.8) +
geom_smooth(method="loess", se=F, span=spanV) +
theme(plot.subtitle=element_text(size=8, hjust=0, color="black")) +
annotate("text", x = fstDay, y = 0.95 * max.newr, label = sprintf("Maximum: %.2f %%", max.newr), hjust = 0, vjust=0,
alpha=0.3, color='steelblue', size=6) +
scale_x_date( labels = date_format("%m/%d"), breaks = "2 weeks") +
xlab(label="") +
ylab(label="%") +
theme(legend.position="none") +
ggtitle(sprintf ("%s: deaths/cases %% (%s)", t0$id, last.obs) )
## Rysunek4: łączne zgony/przypadki *100%
prt0 <- ggplot(d0, aes(x= as.Date(date, format="%Y-%m-%d"), y=totr)) +
geom_point(aes(group = id, color = id), size=.8) +
geom_smooth(method="loess", se=F, span=spanV) +
theme(plot.subtitle=element_text(size=8, hjust=0, color="black")) +
annotate("text", x = fstDay, y = 0.95 * maxRR, label = sprintf("Average: %.2f %%", last.totr), hjust = 0, vjust=0,
alpha=0.3, color='steelblue', size=6) +
scale_x_date( labels = date_format("%m/%d"), breaks = "2 weeks") +
xlab(label="") +
ylab(label="%") +
theme(legend.position="none") +
annotate("text", x = srcDay, y = 0, label = surl, hjust = 1, alpha=.3, size=3) +
ggtitle(sprintf ("%s: total deaths/cases %% (%s)", t0$id, last.obs) )
p00 <- ggarrange(pc0,pd0, pr0, prt0, ncol=2, nrow=2)
ggsave(plot=p00, c0o, width=15)
Użycie:
Rscript --vanilla c19.R --iso PL
albo:
for i in 'AU' 'BR' 'IN' 'US' 'ES' 'SE' 'PL' 'DE' 'GB'; Rscript --vanilla c19.R --iso $i done
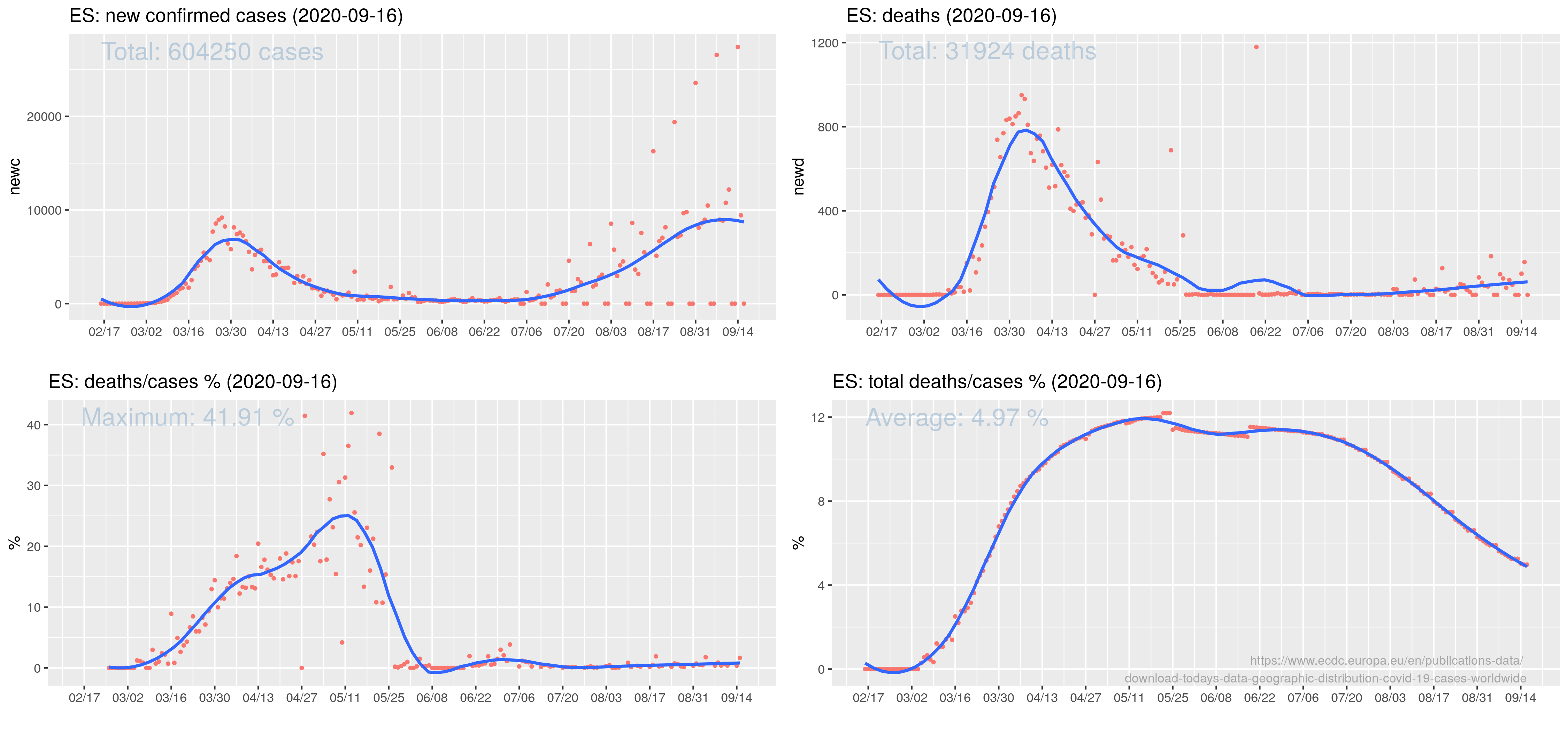
Przykładowy wynik dla Hiszpanii
Jak widać jakość danych jest katastrofalna: pojawiające się liczne zera to w rzeczywistości brak danych. Zwykle sobota/niedziela zero a potem sruuuu 30 tysięcy bo za trzy dni razem. Wszyscy są zmęczeni w tej Hiszpanii pandemią i nawet nie chce im się danych podsyłać do ECDC?
Brak komentarzy:
Prześlij komentarz