Marimekko to zestawiony do 100% wykres słupkowy, gdzie szerokość słupka jest proporcjonalna do jego udziału w liczebności. (https://predictivesolutions.pl/wykres-marimekko-czyli-analityczny-patchwork)
Nie wiem o co chodzi, ale patrząc na przykłady, to jest to wykres pokazujący strukturę dwóch zmiennych na raz. Coś jak skumulowany wykres słupkowy (stacked barchart), tyle że słupki mają zmienną szerokość, odpowiadającą udziałom/liczebnościom wartości jednej cechy. Dla każdego słupka z kolei poszczególne segmenty mają wysokości proporcjonalne do udziałów/liczebności wartości drugiej cechy (w tym słupku). Alternatywną nazwą jest wykres mozaikowy.
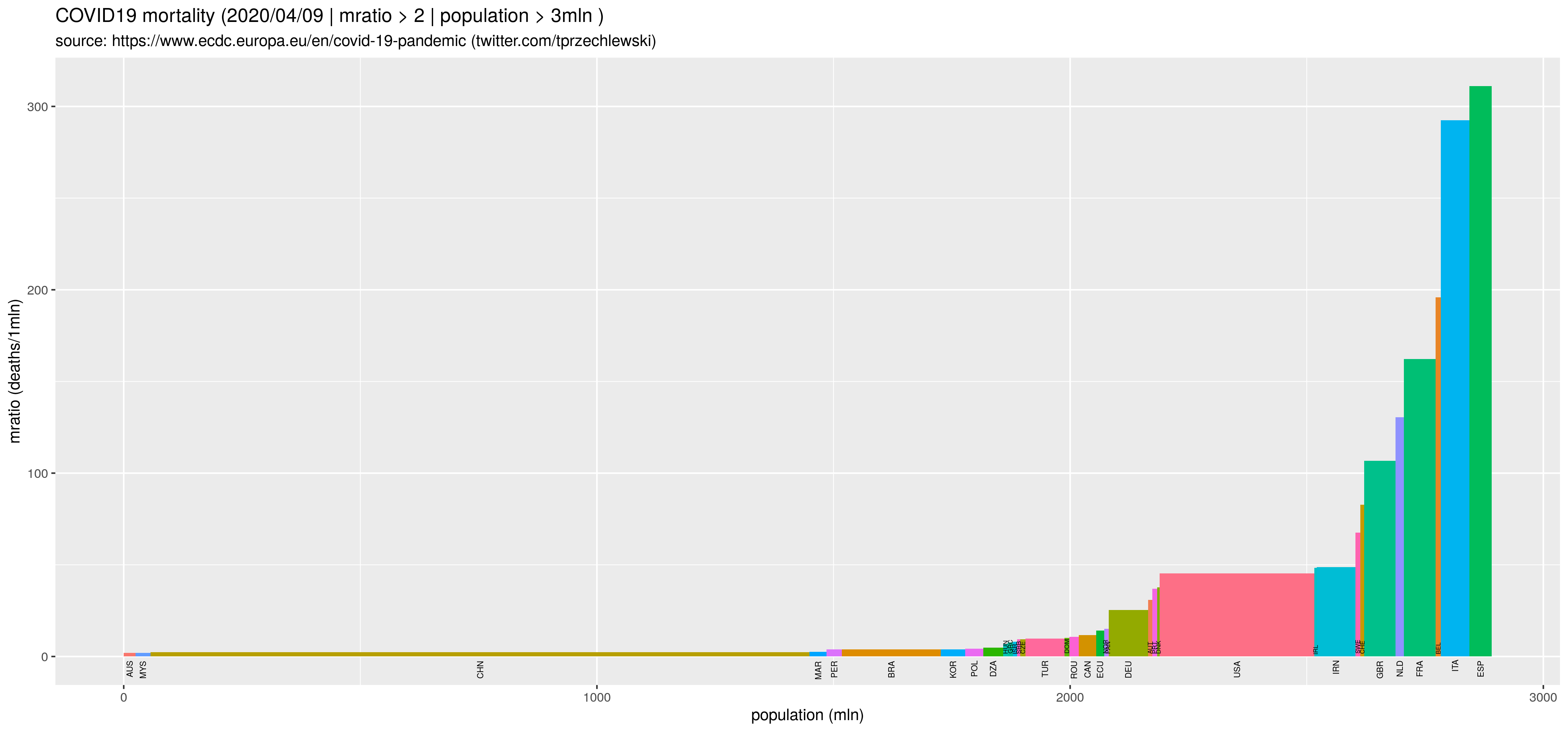
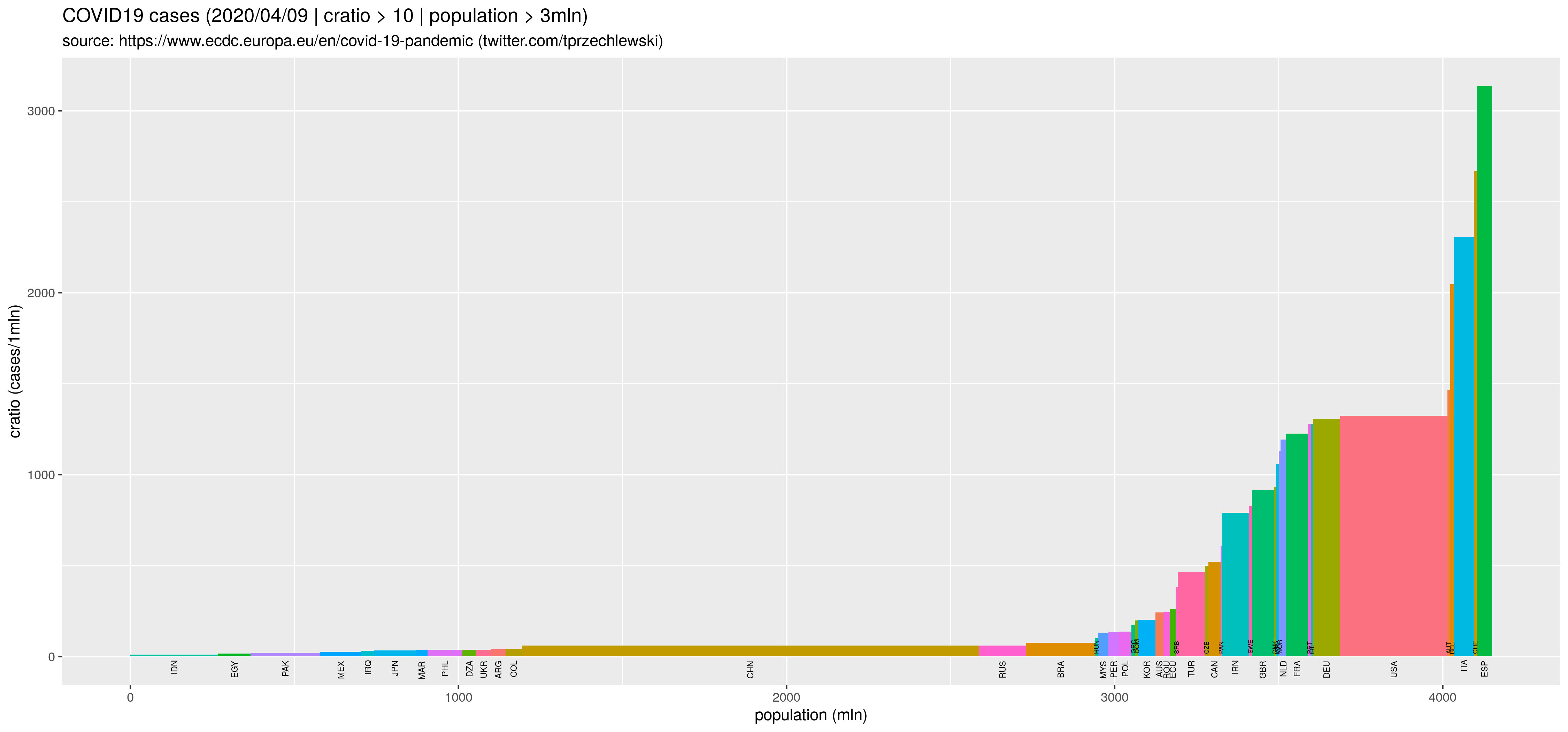
Ale jest też trochę inny wariant takich wykresów, dla przypadku kiedy dla każdej jednostki w populacji generalnej jest Wi = Ci/Ni. Jeżeli wysokość słupka jest proporcjonalna do wartości Wi, szerokość jest proporcjonalna do Ni, to pola są oczywiście w proporcji Ci. Przykładowo jeżeli populacją generalną są kraje świata, wartością cechy Ni jest liczba mieszkańców w kraju i, wartością cechy Ci wielkość emisja CO2 w kraju i, to Wi jest oczywiście emisją per capita.
Moim zdaniem użyteczne, bo pokazuje na raz dwa natężenia: łączne, w skali całej populacji oraz szczegółowe, w skali jednostki. Kontynuując przykład: ile emituje przeciętny mieszkaniec kraju i oraz jaki jest udział emisji kraju i w całości emisji.
W przypadku epidemii COVID19 podstawową zmienną jest liczba zarażonych/zmarłych w kraju i. Ale jeżeli chcemy porównać kraj i z krajem j to oczywiście należy uwzględnić liczbę mieszkańców w obu krajach. Czyli wysokości słupków powinny odpowiadać liczbie zarażonych/zmarłych na jednostkę (np. na 1mln) a szerokości liczbie mieszkańców:
library(ggplot2)
library(dplyr)
dat <- "2020/04/09"
d <- read.csv("indcs.csv", sep = ';', header=T, na.string="NA");
## liczba ludności w milionach (szerokość):
d$popm <- d$pop / million
## Oblicz współczynniki na 1mln
d$casesr <- d$cases/d$popm
### Wysokość:
d$deathsr <- d$deaths/d$popm
## Ograniczamy liczbę krajów żeby zwiększyć czytelność wykresu
## Tylko kraje wykazujące zmarłych
d <- d %>% filter(deaths > 0) %>% as.data.frame
## Tylko kraje z min 2/1mln i populacji > 1mln
d9 <- d %>% filter(deathsr > 2 & popm > 3 & deaths > 49) %>% droplevels() %>%
arrange (deathsr) %>% as.data.frame
d9$w <- cumsum(d9$popm)
d9$wm <- d9$w - d9$popm
d9$wt <- with(d9, wm + (w - wm)/2)
d8$w <- cumsum(d8$popm)
d8$wm <- d8$w - d8$popm
d8$wt <- with(d8, wm + (w - wm)/2)
## Dzielimy etykiety na dwie grupy
## (inaczej wiele etykiet zachodzi na siebie)
d9$iso3h <- d9$iso3
d9$iso3l <- d9$iso3
## Kraje o niskich wartościach bez etykiet
d9$iso3h[ (d9$popm < 15 ) ] <- ""
## Kraje o wysokich wartościach bez etykiet
d9$iso3l[ (d9$popm >= 15 ) ] <- ""
p9 <- ggplot(d9, aes(ymin = 0)) +
ylab(label="mratio (deaths/1mln)") +
xlab(label="population (mln)") +
ggtitle(sprintf("COVID19 mortality (%s | mratio > 2 | population > 3mln )", dat),
subtitle="source: https://www.ecdc.europa.eu/en/covid-19-pandemic (twitter.com/tprzechlewski)") +
geom_rect(aes(xmin = wm, xmax = w, ymax = deathsr, fill = iso3)) +
geom_text(aes(x = wt, y = 0, label = iso3h), vjust=+0.5, hjust=+1.25, size=2.0, angle = 90) +
geom_text(aes(x = wt, y = 0, label = iso3l), vjust=+0.5, hjust=-0.20, size=1.5, angle = 90) +
theme(legend.position = "none")
## ... podobnie dla zmiennej casesr
Wynik w postaci rysunków:
Powyższe jest dostępne tutaj
Brak komentarzy:
Prześlij komentarz