Na stronie https://ourworldindata.org/coronavirus-source-data są dane nt liczby przypadków/zgonów z powodu zarażenia wirusem covid19, których źródłem są 'Raporty Sytuacyjne WHO' (https://www.who.int/emergencies/diseases/novel-coronavirus-2019/situation-reports/). Raporty są w formacie PDF więc bezpośrednio nie można korzystać z publikowanych tam danych. No ale uprzejmi ludzie z ourworldindata.org już te raporty zamienili na csv i są one gotowe do pobrania:
wget -N https://covid.ourworldindata.org/data/full_data.csv
Za pomocą prostych skryptów Perla modyfikuję plik full_data.csv tak, żeby poszczególne kolumny zawierały: date;id;country;newc;newd;totalc;totald (data, ISO-kod kraju, nazwa-kraju, nowe-przypadki, nowe-zgony, wszystkie-przypadki, wszystkie-zgony)
Dla wybranych krajów rysują wykresy liniowe (wykorzystując R):
library("dplyr")
library("ggplot2")
library("ggpubr")
##
today <- Sys.Date()
tt<- format(today, "%d/%m/%Y")
d <- read.csv("covid19.csv", sep = ';', header=T, na.string="NA");
d <- d %>% filter(as.Date(date, format="%Y-%m-%d") > "2020-02-15") %>% as.data.frame
##
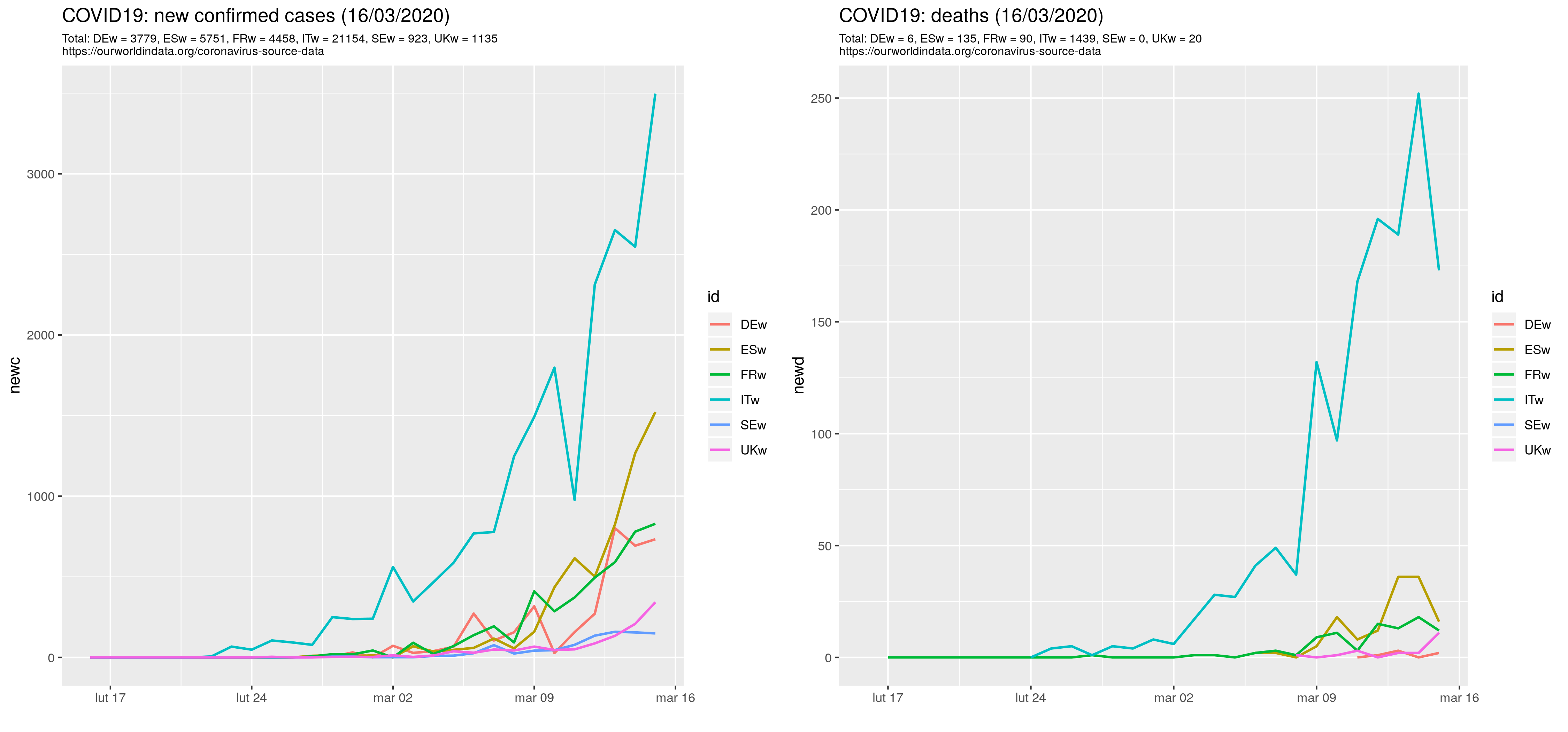
c1 <- c('ITw', 'DEw', 'ESw', 'UKw', 'FRw', 'DKw', 'SEw')
# date;id;country;newc;newd;totalc;totald
d1 <- d %>% filter (id %in% c1) %>% as.data.frame
t1 <- d1 %>% group_by(id) %>% summarise(cc = sum(newc, na.rm=T), dd=sum(newd, na.rm=T))
lab1c <- toString(paste (sep=" = ", t1$id, t1$cc))
lab1d <- toString(paste (sep=" = ", t1$id, t1$dd))
str(d1)
pc1 <- ggplot(d1, aes(x= as.Date(date, format="%Y-%m-%d"), y=newc)) + geom_line(aes(group = id, color = id), size=.8) +
xlab(label="") +
theme(plot.subtitle=element_text(size=8, hjust=0, color="black")) +
ggtitle(sprintf("COVID19: new confirmed cases (%s)", tt), subtitle=sprintf("Total: %s\n%s", lab1c, surl))
pd1 <- ggplot(d1, aes(x= as.Date(date, format="%Y-%m-%d"), y=newd)) + geom_line(aes(group = id, color = id), size=.8) +
xlab(label="") +
theme(plot.subtitle=element_text(size=8, hjust=0, color="black")) +
ggtitle(sprintf ("COVID19: deaths (%s)", tt), subtitle=sprintf("Total: %s\n%s", lab1d, surl))
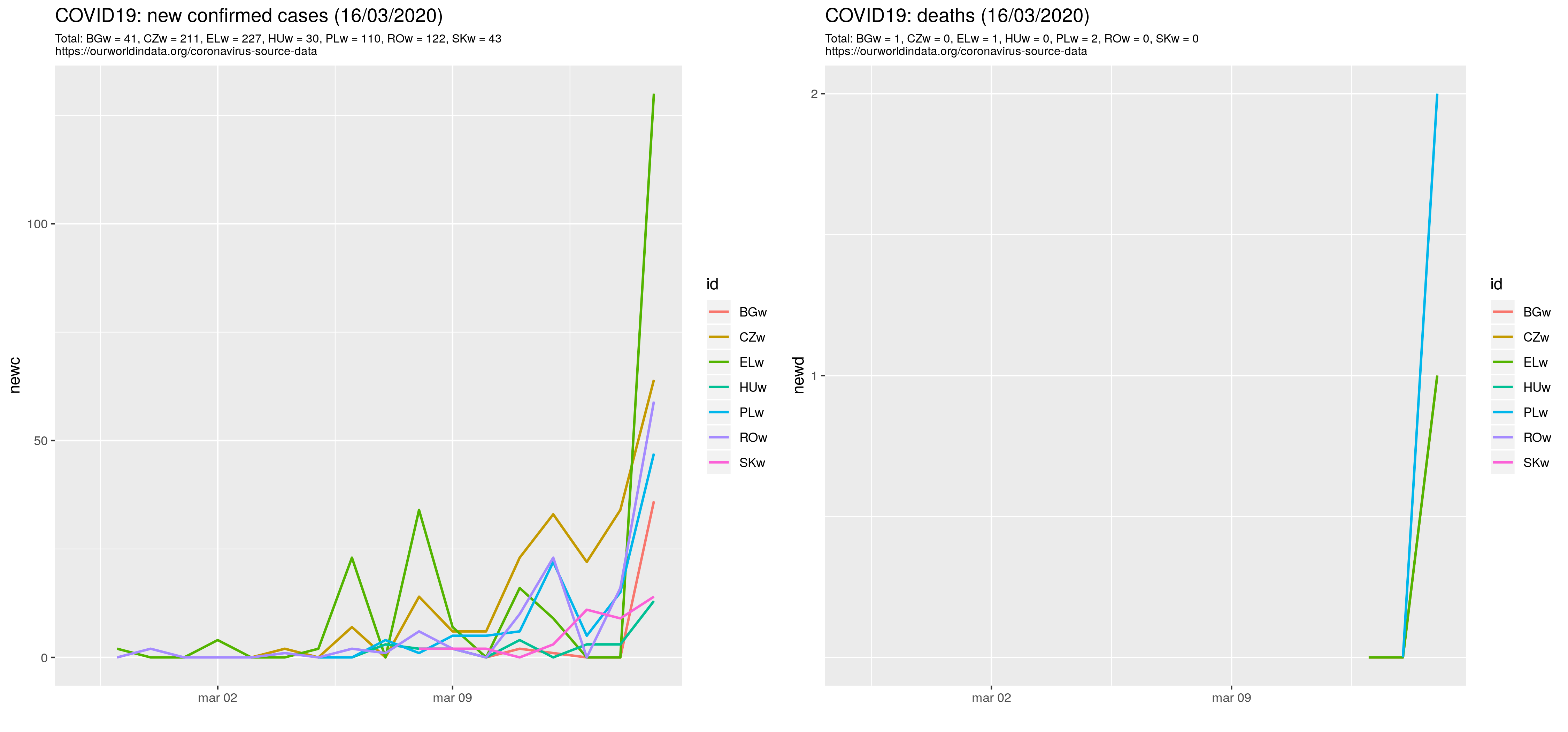
c2 <- c('PLw', 'CZw', 'SKw', 'HUw', 'ROw', 'BGw', 'ELw')
d2 <- d %>% filter (id %in% c2) %>% as.data.frame
t2 <- d2 %>% group_by(id) %>% summarise(cc = sum(newc, na.rm=T), dd=sum(newd, na.rm=T))
str(d2)
lab2c <- toString(paste (sep=" = ", t2$id, t2$cc))
lab2d <- toString(paste (sep=" = ", t2$id, t2$dd))
pc2 <- ggplot(d2, aes(x= as.Date(date, format="%Y-%m-%d"), y=newc)) + geom_line(aes(group = id, color = id), size=.8) +
theme(plot.subtitle=element_text(size=8, hjust=0, color="black")) +
xlab(label="") +
ggtitle(sprintf("COVID19: new confirmed cases (%s)", tt), subtitle=sprintf("Total: %s\n%s", lab2c, surl))
pd2 <- ggplot(d2, aes(x= as.Date(date, format="%Y-%m-%d"), y=newd)) + geom_line(aes(group = id, color = id), size=.8) +
theme(plot.subtitle=element_text(size=8, hjust=0, color="black")) +
xlab(label="") +
scale_y_continuous(breaks=c(1,2,3,4,5,6,7,8,9)) +
ggtitle(sprintf ("COVID19: deaths (%s)", tt), subtitle=sprintf("Total: %s\n%s", lab2d, surl))
p1 <- ggarrange(pc1,pd1, ncol=2, nrow=1)
p2 <- ggarrange(pc2,pd2, ncol=2, nrow=1)
ggsave(plot=p1, "Covid19_1w.png", width=15)
ggsave(plot=p2, "Covid19_2w.png", width=15)
Zatem: Liczba przypadków/zgonów z powodu zarażenia wirusem covid19 na podstawie danych ourworldindata.org/WHO:
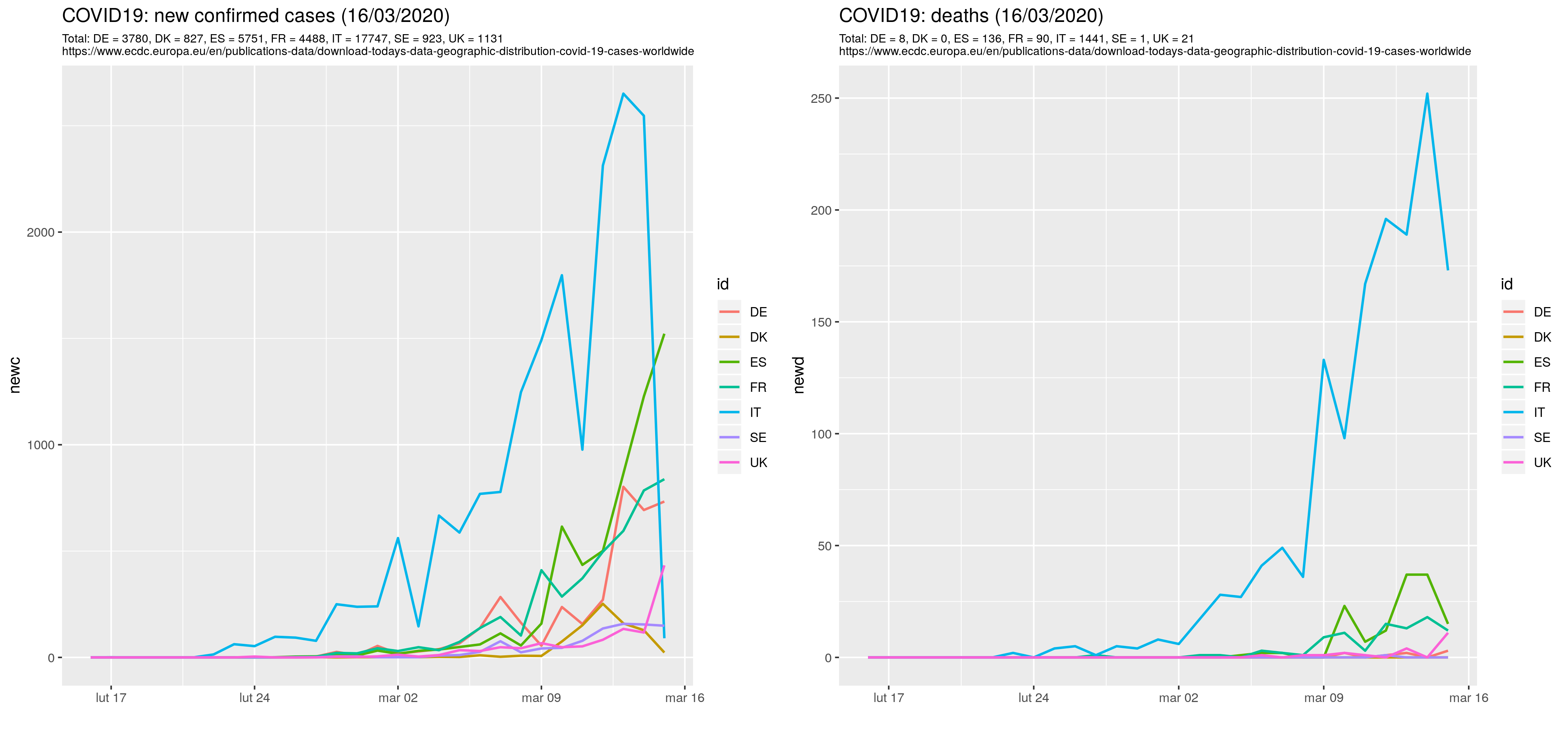
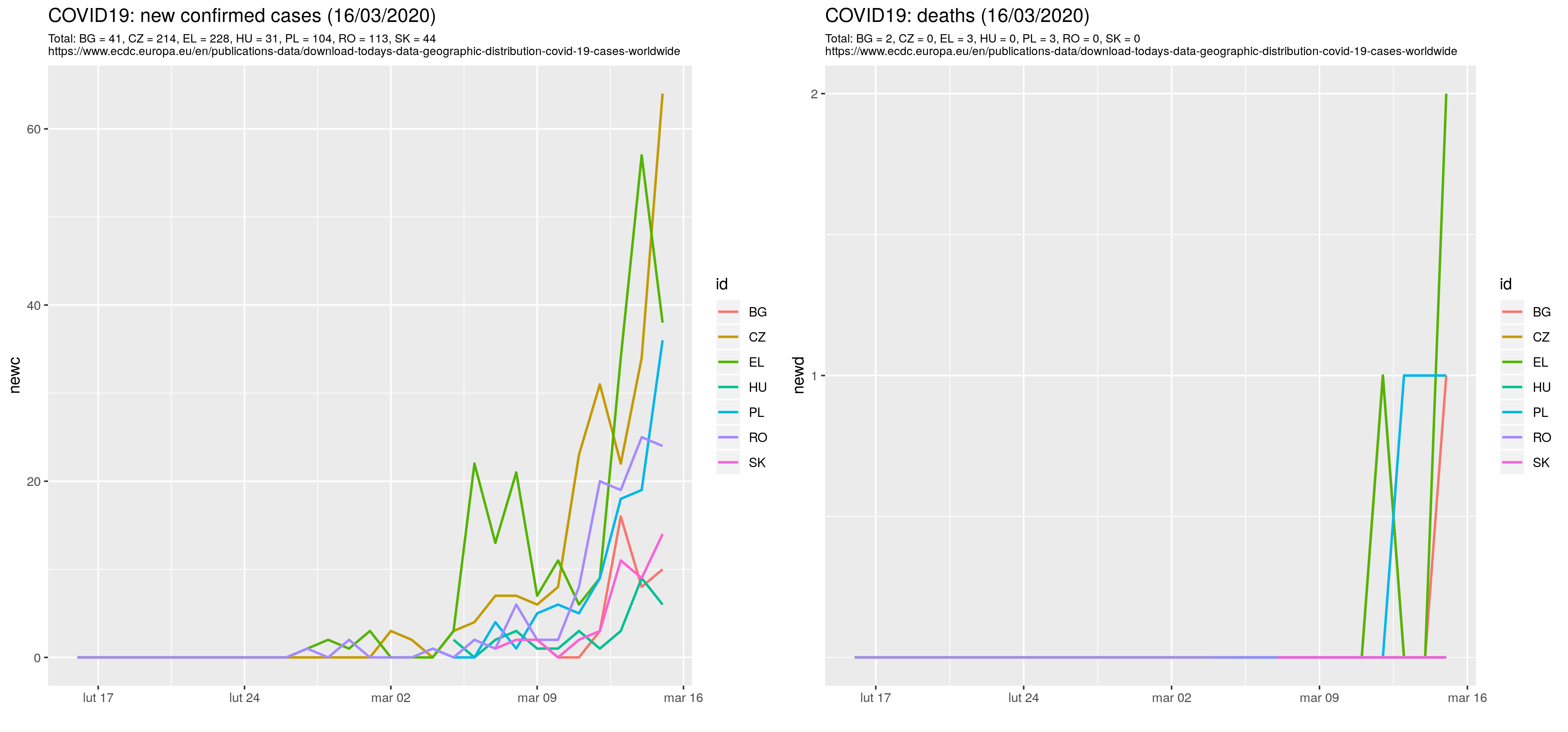
UE, która jest powszechnie krytykowana, że nic nie robi w sprawie, okazuje się że coś tobi -- też udostępnia jakieś dane w temacie (https://www.ecdc.europa.eu/en/publications-data/download-todays-data-geographic-distribution-covid-19-cases-worldwide). Wprawdzie nie jest określone skąd te dane pochodzą, ale sądząc po ich zawartości źródło jest to samo (WHO).
wget -N https://www.ecdc.europa.eu/sites/default/files/documents/COVID-19-geographic-disbtribution-worldwide-2020-03-15.xls\ -O covid19.csv
Ponieważ dane, że tak powiem, unijne są w formacie xls zamieniam je na csv, wykorzystując do tego LibreOffice:
## zamień wszystkie pliki z bieżącego katalogu na csv ze ; (59) jako znakiem separacji: soffice --convert-to csv:"Text - txt - csv (StarCalc)":59,,0,1,1 --outdir . *.xls
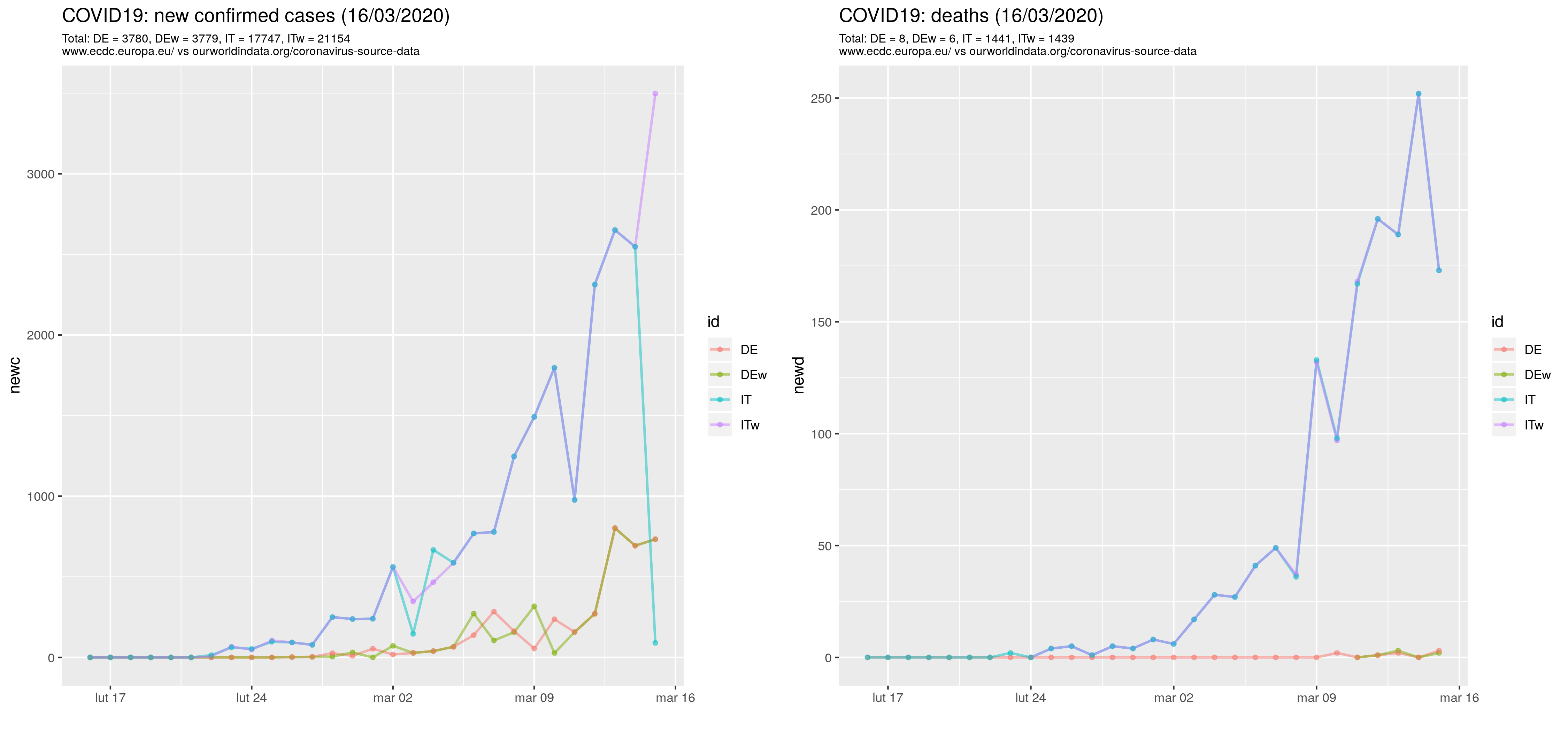
Rysuję wykresy liniowe zmodyfikowanym z dokładnością do pliku z danymi R-skryptem. Wyniki są prawie takie same. Może bym nawet nie zwrócił uwagi, że się różnią gdyby nie podejrzane załamanie liczby przypadków dla Włoch dla 15.03.2020 (z 2,5 tys na 90).
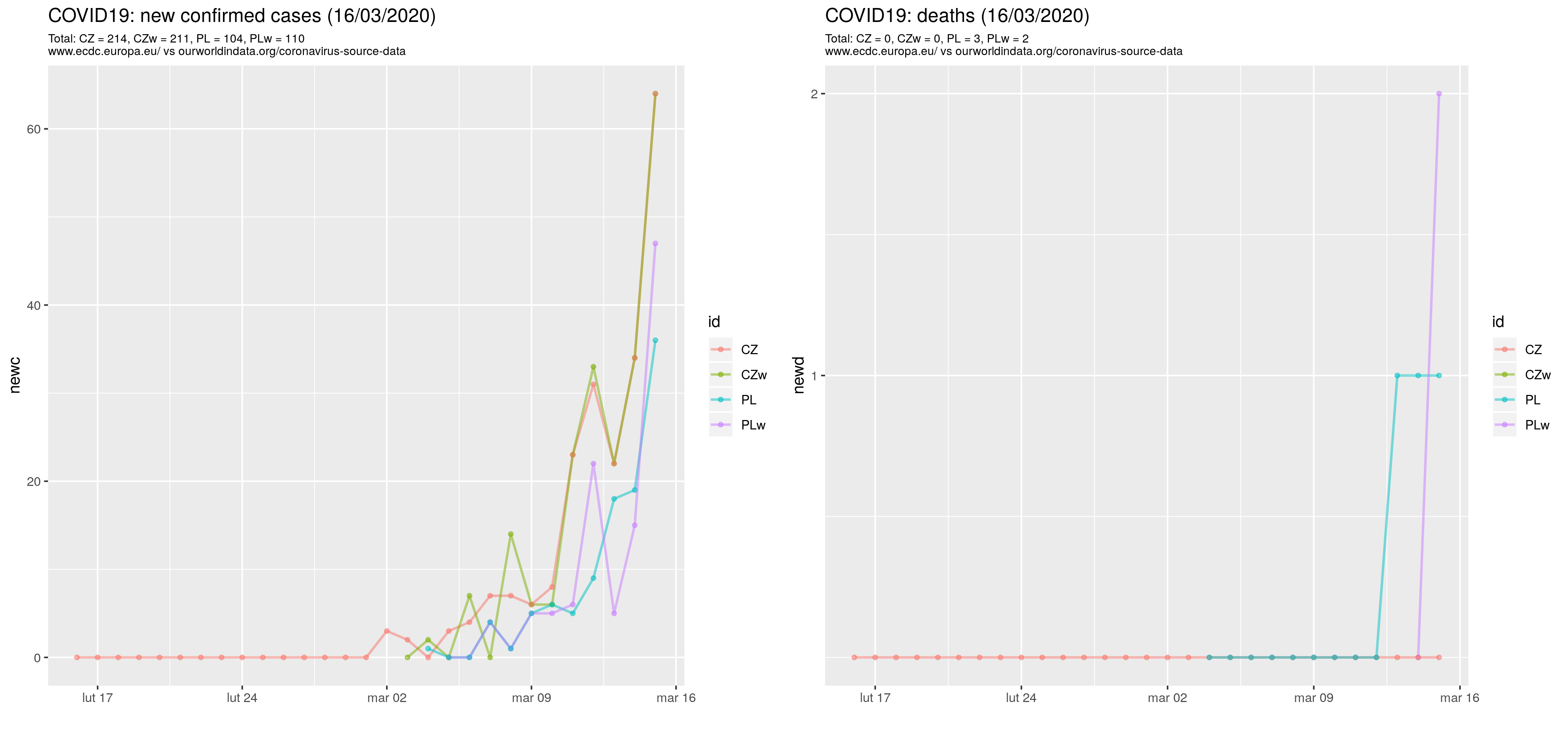
Drążąc temat wyrysowałem wykresy dla wybranych czterech krajów w dwóch wariantach danych (dane z ourworldindata.org oznaczone literką w). W szczególności i niestety Włochy 15/3/2020 odnotowały ponad 3497 nowych przypadków a nie 90 jak podano w bazie Unijnej. Są też mniejsze różnice w innych miejscach:
Wszystko to robione jest automatem co pobiera/zamienia/rysuje/wstawia na githuba (https://github.com/hrpunio/Nafisa/tree/master/Covid19) oraz wysyła na twittera (https://twitter.com/tprzechlewski). Automat działa na RaspberryPi zresztą...
Brak komentarzy:
Prześlij komentarz