MZ to Ministerstwo Zdrowia. Specyfiką PL jest brak danych publicznych nt. Pandemii.. Na stronie GIS nie ma nic, a stacje wojewódzkie publikują jak chcą i co chcą. Ministerstwo zdrowia z kolei na swojej stronie podaje tylko dane na bieżący dzień, zaś na amerykańskim Twitterze publikuje komunikaty na temat. To co jest na stronie znika oczywiście następnego dnia zastępowane przez inne cyferki. Do tego są tam tylko dzienne dane zbiorcze o liczbie zarażonych i zmarłych, (w podziale na województwa). Nie ma na przykład wieku, płci itp... To co jest na Twitterze z kolei ma formę tekstowego komunikatu postaci: Mamy 502 nowe i potwierdzone przypadki zakażenia #koronawirus z województw: małopolskiego (79), śląskiego (77), mazowieckiego (75)... [...] Z przykrością informujemy o śmierci 6 osób zakażonych koronawirusem (wiek-płeć, miejsce zgonu): 73-K Kędzierzyn-Koźle, 75-M Łańcut, 92-K Lipie, 72-M, 87-M, 85-K Gdańsk.
Czyli podają zarażonych ogółem i w podziale na województwa oraz dane indywidualne zmarłych w postaci płci i wieku oraz miejsca zgonu (miasta żeby było inaczej niż w przypadku podawania zakażeń.)
No więc chcę wydłubać dane postaci 92-K z tweetów publikowanych przez Ministerstwo Zdrowia. W tym celu za pomocą tweepy pobieram cały streamline (+3200 tweetów zaczynających się jeszcze w 2019 roku), ale dalej to już działam w Perlu, bo w Pythonie jakby mniej komfortowo się czuję. Po pierwsze zamieniam format json na csv:
use JSON;
use Data::Dumper;
use Time::Piece;
use open ":encoding(utf8)";
use open IN => ":encoding(utf8)", OUT => ":utf8";
binmode(STDOUT, ":utf8");
## ID = tweeta ID; date = data;
## repid -- odpowiedź na tweeta o numerze ID
## text -- tekst tweeta
print "id;date;repid;text\n";
while (<>) {
chomp();
$tweet = $_;
my $json = decode_json( $tweet );
#print Dumper($json);
$tid = $json->{"id"};
$dat = $json->{"created_at"};
## Data jest w formacie rozwlekłym zamieniamy na YYYY-MM-DDTHH:MM:SS
## Fri Oct 04 14:48:25 +0000 2019
$dat = Time::Piece->strptime($dat,
"%a %b %d %H:%M:%S %z %Y")->strftime("%Y-%m-%dT%H:%M:%S");
$rep = $json->{"in_reply_to_status_id"};
$ttx = $json->{"full_text"}; $ttx =~ s/\n/ /g;
## Zamieniamy ; na , w tekście żeby użyć ; jako separatora
$ttx =~ s/;/,/g; ####
print "$tid;$dat;$rep;$ttx\n";
Komunikaty dłuższe niż limit Twittera są dzielone na kawałki, z których każdy jest odpowiedzią na poprzedni, np:
1298900644267544576;2020-08-27T08:30:20;1298900642522685440;53-M, 78-K i 84-K Kraków. Większość osób ... 1298900642522685440;2020-08-27T08:30:20;1298900640714948608;67-K Lublin (mieszkanka woj. podkarpackiego), 85-K Łańcut,... 1298900640714948608;2020-08-27T08:30:19;1298900639586680833;kujawsko-pomorskiego (24), świętokrzyskiego (18), opolskiego... 1298900639586680833;2020-08-27T08:30:19;;Mamy 887 nowych i potwierdzonych przypadków zakażenia #koronawirus z województw: ...
Czyli tweet 1298900639586680833 zaczyna, 1298900640714948608 jest odpowiedzią na 1298900639586680833, a 1298900642522685440 odpowiedzią na 1298900640714948608 itd. Tak to sobie wymyślili... W sumie chyba niepotrzebnie, ale w pierwszym kroku agreguję podzielone komunikaty w ten sposób, że wszystkie odpowiedzi są dołączane do pierwszego tweeta (tego z pustym polem in_reply_to_status_id):
## nextRef jest rekurencyjna zwraca numer-tweeta,
## który jest początkiem wątku
sub nextRef {
my $i = shift;
if ( $RR{"$i"} > 0 ) {
return ( nextRef( "$RR{$i}" ) );
} else { return "$i" }
}
### ### ###
while (<>) { chomp();
($id, $d, $r, $t) = split /;/, $_;
$TT{$id} = $t;
$RR{$id} = $r;
$DD{$id} = $d;
}
### ### ###
for $id ( sort keys %TT ) {
$lastId = nextRef("$id");
$LL{"$id"} = $lastId;
$LLIds{"$lastId"} = "$lastId";
}
### ### ###
for $id (sort keys %TT) {
## print "### $DD{$id};$id;$LL{$id};$TT{$id}\n"; }
$TTX{$LL{"$id"}} .= " " . $TT{"$id"};
$DDX{$LL{"$id"}} .= ";" . $DD{"$id"};
}
### ### ###
for $i (sort keys %TTX) {
$dates = $DDX{"$i"};
$dates =~ s/^;//; ## pierwszy ; jest nadmiarowy
@tmpDat = split /;/, $dates;
$dat_time_ = $tmpDat[0];
($dat_, $time_) = split /T/, $dat_time_;
$ffN = $#tmpDat + 1;
$collapsedTweet = $TTX{$i};
print "$i;$dat_;$time_;$ffN;$collapsedTweet\n";
}
Zapuszczenie powyższego powoduje konsolidację wątków, tj. np. powyższe 4 tweety z 2020-08-27 połączyły się w jeden:
1298900639586680833;2020-08-27;08:30:19;4; Mamy 887 nowych i potwierdzonych przypadków zakażenia #koronawirus z województw: małopolskiego (233), śląskiego (118), mazowieckiego (107), [...] Liczba zakażonych koronawirusem: 64 689 /2 010 (wszystkie pozytywne przypadki/w tym osoby zmarłe).
Teraz wydłubujemy tylko tweety z frazą nowych i potwierdzonych albo nowe i potwierdzone:
## nowych i potwierdzonych albo nowe i potwierdzone ## (MZ_09.csv to CSV ze `skonsolidowanymi' wątkami) cat MZ_09.csv | grep 'nowych i pot\|nowe i pot' > MZ_09_C19.csv wc -l MZ_09_C19.csv 189 MZ_09_C19.csv
Wydłubanie fraz wiek-płeć ze skonsolidowanych wątków jest teraz proste:
perl -e '
while (<>) {
($i, $d, $t, $c, $t) = split /;/, $_;
while ($t =~ m/([0-9]+-[MK])/g ) {
($w, $p) = split /\-/, $1;
print "$d;$w;$p\n";
}
}' MZ_09_C19.csv > C19D.csv
wc -l C19PL_down.csv
1738
Plik wykazał 1738 osób. Pierwsza komunikat jest z 16 kwietnia. Ostatni z 31. sierpnia. Pierwsze zarejestrowane zgony w PL odnotowano 12 marca (albo 13 nieważne). W okresie 12 marca --15 kwietnia zmarło 286 osób. Dodając 286 do 1738 wychodzi 2024. Wg MZ w okresie 12.03--31.08 zmarło 2039. Czyli manko wielkości 15 zgonów (około 0,5%). No trudno, nie chce mi się dociekać kto i kiedy pogubił tych 15...
Równie prostym skryptem zamieniam dane indywidualne na tygodniowe
#!/usr/bin/perl -w
use Date::Calc qw(Week_Number);
while (<>) {
chomp();
##if ($_ =~ /age/) { next } ##
my ($d, $w, $p ) = split /;/, $_;
my ($y_, $m_, $d_) = split /\-/, $d;
my $week = Week_Number($y_, $m_, $d_);
$DW{$week} += $w;
$DN{$week}++;
$DD{$d} = $week;
$YY{$week} = 0;
$YY_last_day{$week} = $d;
## wiek wg płci
$PW{$p} += $w;
$PN{$p}++;
}
for $d (keys %DD) {
$YY{"$DD{$d}"}++; ## ile dni w tygodniu
}
print STDERR "Wg płci/wieku (ogółem)\n";
for $p (keys %PW) {
$s = $PW{$p}/$PN{$p};
printf STDERR "%s %.2f %i\n", $p, $s, $PN{$p};
$total += $PN{$p};
}
print STDERR "Razem: $total\n";
print "week;deaths;age;days;date\n";
for $d (sort keys %DW) {
if ($YY{$d} > 2 ) {## co najmniej 3 dni
$s = $DW{$d}/$DN{$d};
printf "%s;%i;%.2f;%i;%s\n", $d, $DN{$d}, $s, $YY{$d}, $YY_last_day{$d};
}
}
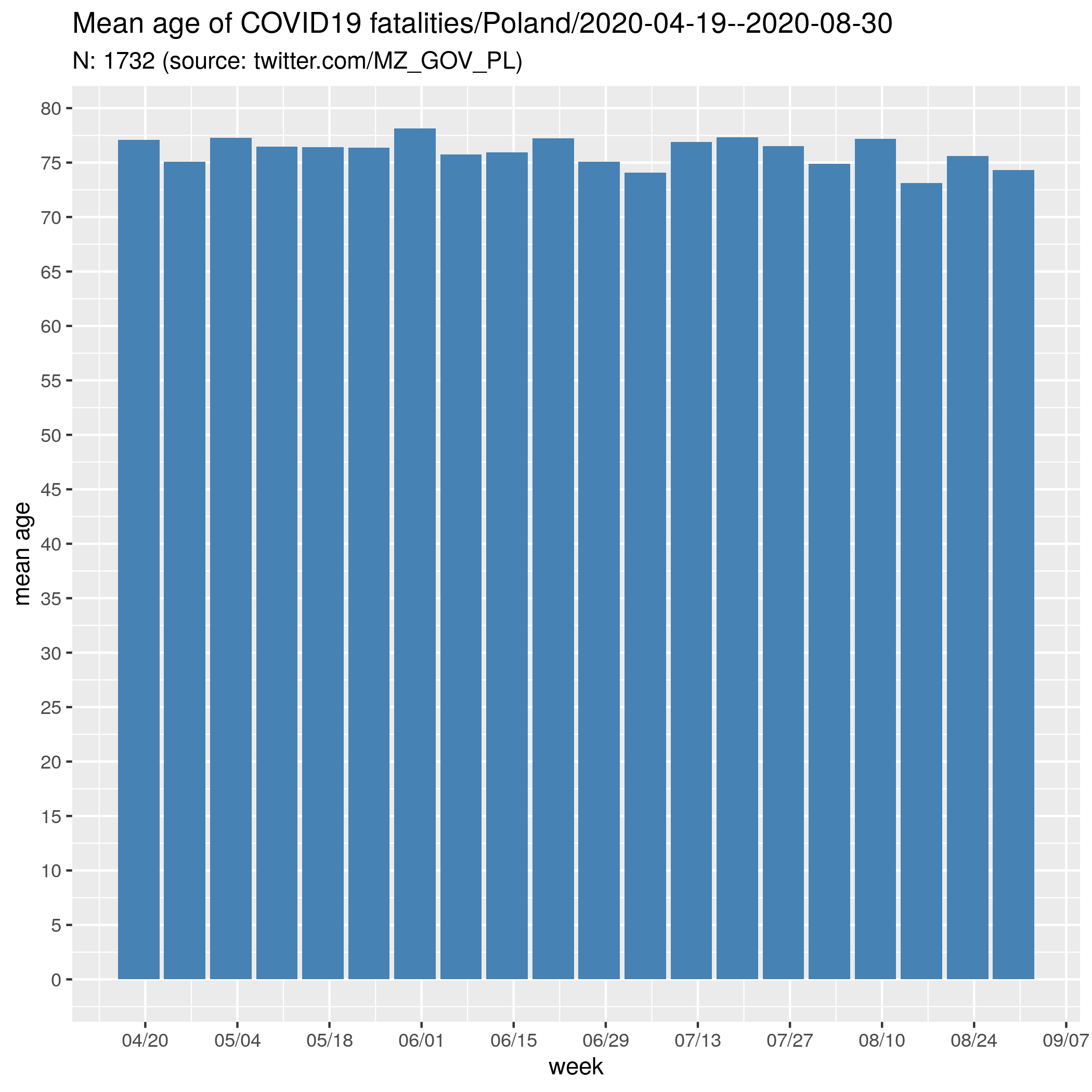
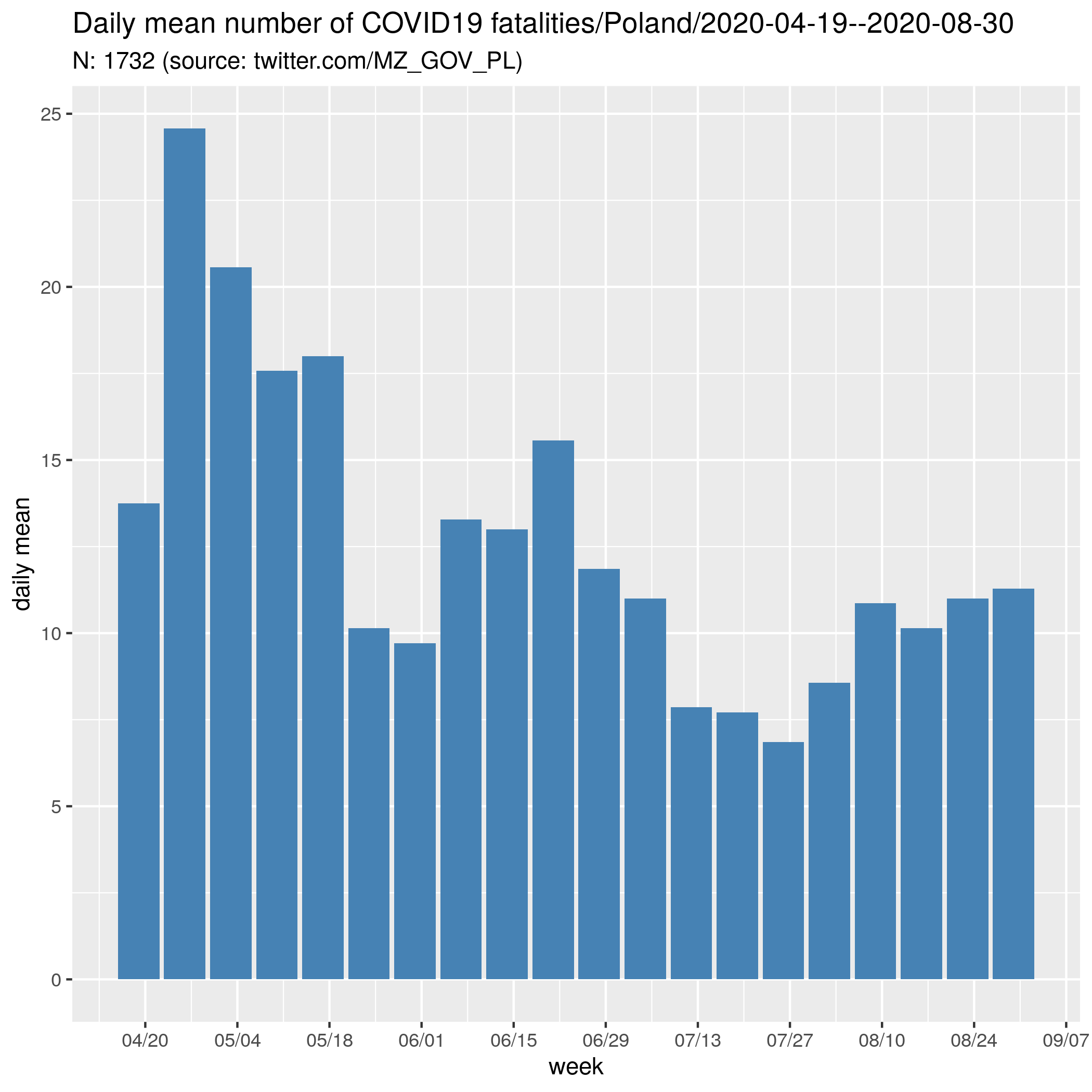
Co daje ostatecznie (week -- numer tygodnia w roku; meanage -- średni wiek zmarłych; deaths -- liczba zmarłych w tygodniu; days -- dni w tygodniu; date -- ostatni dzień tygodnia):
week;deaths;meanage;days;date 16;55;77.07;4;2020-04-19 17;172;75.09;7;2020-04-26 18;144;77.29;7;2020-05-03 19;123;76.46;7;2020-05-10 20;126;76.40;7;2020-05-17 21;71;76.37;7;2020-05-24 22;68;78.12;7;2020-05-31 23;93;75.73;7;2020-06-07 24;91;75.93;7;2020-06-14 25;109;77.24;7;2020-06-21 26;83;75.06;7;2020-06-28 27;77;74.09;7;2020-07-05 28;55;76.91;7;2020-07-12 29;54;77.33;7;2020-07-19 30;48;76.52;7;2020-07-26 31;60;74.88;7;2020-08-02 32;76;77.17;7;2020-08-09 33;71;73.11;7;2020-08-16 34;77;75.61;7;2020-08-23 35;79;74.33;7;2020-08-30
To już można na wykresie przedstawić:-)
library("dplyr")
library("ggplot2")
library("scales")
##
spanV <- 0.25
d <- read.csv("C19D_weekly.csv", sep = ';', header=T, na.string="NA")
first <- first(d$date)
last <- last(d$date)
period <- sprintf ("%s--%s", first, last)
d$deaths.dailymean <- d$deaths/d$days
cases <- sum(d$deaths);
max.cases <- max(d$deaths)
note <- sprintf ("N: %i (source: twitter.com/MZ_GOV_PL)", cases)
pf <- ggplot(d, aes(x= as.Date(date), y=meanage)) +
geom_bar(position="dodge", stat="identity", fill="steelblue") +
scale_x_date( labels = date_format("%m/%d"), breaks = "2 weeks") +
scale_y_continuous(breaks=c(0,5,10,15,20,25,30,35,40,45,50,55,60,65,70,75,80)) +
xlab(label="week") +
ylab(label="mean age") +
ggtitle(sprintf ("Mean age of COVID19 fatalities/Poland/%s", period), subtitle=note )
note <- sprintf ("N: %i (source: twitter.com/MZ_GOV_PL)", cases)
pg <- ggplot(d, aes(x= as.Date(date), y=deaths.dailymean)) +
geom_bar(position="dodge", stat="identity", fill="steelblue") +
scale_x_date( labels = date_format("%m/%d"), breaks = "2 weeks") +
scale_y_continuous(breaks=c(0,5,10,15,20,25,30,35,40,45,50)) +
xlab(label="week") +
ylab(label="daily mean") +
ggtitle(sprintf("Daily mean number of COVID19 fatalities/Poland/%s", period), subtitle=note )
ggsave(plot=pf, file="c19dMA_w.png")
ggsave(plot=pg, file="c19dN_w.png")
Wynik tutaj:
Średnia Wg płci/wieku/ogółem. Ogółem -- 75,9 lat (1738 zgonów) w tym: mężczyźni (914 zgonów) -- 73.9 lat oraz kobiety (824) -- 78.4 lat. Stan pandemii na 31.08 przypominam. Apogeum 2 fali... Średnia wieku w PL (2019 rok) 74,1 lat (M) oraz 81,8 lat (K).
Świetny wpis. Nawet nie wiedziałam że można w ten sposób odczytać wiele informacji.
OdpowiedzUsuń